adenilazione - Adenylylation
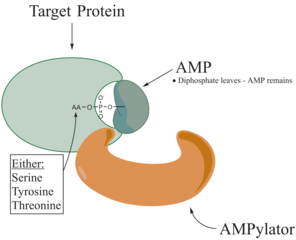
L'adenililazione , più comunemente nota come AMPilazione , è un processo in cui una molecola di adenosina monofosfato (AMP) è legata in modo covalente alla catena laterale dell'amminoacido di una proteina . Questa aggiunta covalente di AMP a una catena laterale ossidrile della proteina è una modifica post-traduzionale . L'adenililazione comporta un legame fosfodiestere tra un gruppo ossidrile della molecola in fase di adenililazione e il gruppo fosfato del nucleotide adenosina monofosfato (cioè l'acido adenilico). Gli enzimi in grado di catalizzare questo processo sono chiamati AMPilatori.
Gli amminoacidi noti per essere presi di mira nella proteina sono tirosina e treonina e talvolta serina. Quando le cariche su una proteina subiscono un cambiamento, influenza le caratteristiche della proteina, normalmente alterandone la forma attraverso le interazioni degli amminoacidi che compongono la proteina. L'AMPilazione può avere vari effetti sulla proteina. Queste sono proprietà della proteina come, stabilità, attività enzimatica, legame di cofattori e molte altre capacità funzionali di una proteina. Un'altra funzione dell'adenililazione è l'attivazione degli amminoacidi, che è catalizzata dalla tRNA aminoacil sintetasi. Le proteine più comunemente identificate per ricevere l'AMPilazione sono le GTPasi e la glutammina sintetasi .
Adenilatori
Gli enzimi responsabili dell'AMPilazione, chiamati AMPilatori o Adeniltransferasi , rientrano in due diverse famiglie, tutte a seconda delle loro proprietà strutturali e del meccanismo utilizzato. AMPylator è formato da due metà catalitiche omologhe. Una metà è responsabile della catalizzazione della reazione di adenililazione, mentre l'altra metà catalizza la reazione di deadenililazione fosforolitica . Queste due famiglie sono la DNA- β -polimerasi-like e la famiglia Fic.
DNA- β -polimerasi-like, è una famiglia di Nucleotidyltransferase . Più specificamente è conosciuta come la famiglia GlnE. C'è un motivo specifico che viene utilizzato per chiarire questa particolare famiglia. Il motivo consiste in un foglio β a tre filamenti che fa parte della coordinazione degli ioni magnesio e del legame con il fosfato. L'aspartato è essenziale affinché l'attività avvenga in questa famiglia.
Il dominio Fic appartiene alla superfamiglia Fido (Fic/Doc) La famiglia Fic , che è una filamentazione indotta dal dominio AMP ciclico, è nota per eseguire l'AMPilazione. Questa famiglia di proteine si trova in tutti i domini della vita sulla terra. È mediato da un meccanismo dell'alfa elica del sito di legame dell'ATP. I batteri infettivi utilizzano questo dominio per interrompere la fagocitosi e causare la morte cellulare. I domini Fic sono domini evolutivamente conservati nei procarioti e negli eucarioti che appartengono alla superfamiglia del dominio Fido.
È stato dimostrato che gli AMPilatori sono paragonabili alle chinasi a causa della loro attività di idrolisi dell'ATP e del trasferimento reversibile del metabolita a una catena laterale ossidrile del substrato proteico. Tuttavia, l'AMPilazione catalizza un attacco nucleofilo sul gruppo α-fosfato, mentre la chinasi nella reazione di fosforilazione prende di mira il γ-fosfato. L'attacco nucleofilo dell'AMPilazione porta al rilascio di pirofosfato e la proteina modificata con AMP è il prodotto della reazione di AMPilazione.
De-adenililatori
La de-AMPilazione è la reazione inversa in cui la molecola di AMP viene staccata dal lato amminoacidico di una catena proteica.
Ci sono tre meccanismi noti per questa reazione. La GS-ATase batterica (GlnE) codifica per una proteina bipartita con domini separati di AMPilazione N-terminale e de-AMPilazione C-terminale la cui attività è regolata da P II e da modificazioni post-traduzionali associate. De-AMPilazione del suo substrato La glutammina sintetasi AMPilata procede mediante una reazione fosforolitica tra l'adenil-tirosina di GS e l' ortofosfato , portando alla formazione di ADP e glutammina sintetasi non modificata.
SidD, una proteina introdotta nella cellula ospite dai batteri patogeni Legionella pneumophila, de-AMPylate Rab1, una proteina ospite AMPilata da un diverso enzima della Legionella pneumophila, l'AMPylase SidM. Mentre il vantaggio per il patogeno dell'introduzione di questi due effettori antagonisti nell'ospite rimane poco chiaro, la reazione biochimica condotta da SidD prevede l'uso di un dominio simile alla fosfatasi per catalizzare la rimozione idrolitica dell'AMP dalla tirosina 77 del Rab1 dell'ospite.
Nelle cellule animali la rimozione dell'AMP dalla treonina 518 di BiP/Grp78 è catalizzata dallo stesso enzima, FICD, che AMPila il BiP. A differenza della GS-ATasi batterica, la FICD effettua entrambe le reazioni con lo stesso dominio catalitico.
procarioti
Omeostasi batterica
L'AMPilazione è coinvolta nell'omeostasi batterica. L'esempio più famoso è l'AMPylator GS-ATase (GlnE), che contribuisce alla complessa regolazione del metabolismo dell'azoto attraverso l'AMPilazione della glutammina sintetasi che è stata introdotta nelle parti di AMPilazione e DeAMPilazione.
Un altro esempio di AMPilatori che svolgono un ruolo nell'omeostasi batterica sono gli AMPilatori Fic di classe I (FicT), che modificano la subunità GyrB della DNA girasi, il residuo di tirosina conservato per il legame dell'ATP della subunità ParE alla topoisomerasi IV. Questa inattivazione della DNA girasi mediante AMPilazione porta all'attivazione della risposta SOS, che è la risposta cellulare al danno del DNA. L'attività di FicT AMPylation è reversibile e porta solo all'arresto della crescita, ma non alla morte cellulare. Pertanto, FicT AMPylation svolge un ruolo nella regolazione dello stress cellulare, che è dimostrato nei batteri Wolbachia che il livello di FicT aumenta in risposta alla doxiciclina.
Si trova anche che un AMPylator NmFic di N. meningtidis di classe III modifica l'AMPylate GyrB alla tirosina conservata per il legame dell'ATP. Ciò dimostra che i domini Fic sono altamente conservati, il che indica l'importante ruolo dell'AMPilazione nella regolazione dello stress cellulare nei batteri. La regolazione di NmFic comporta la monomerizzazione concentrazione-dipendente e l'autoAMPilazione per l'attivazione dell'attività di NmFic.
patogenicità batterica
È stato dimostrato che le proteine dei batteri, note anche come effettori, utilizzano l'AMPilazione. Effettori come VopS, IbpA e DrrA hanno dimostrato di AMPylare le GTPasi dell'ospite e causare cambiamenti nel citoscheletro di actina. Le GTPasi sono bersagli comuni degli AMPylator. Le famiglie Rho , Rab e Arf GTPasi sono coinvolte nella dinamica del citoscheletro di actina e nel traffico vescicolare. Svolgono anche ruoli nei meccanismi di controllo cellulare come la fagocitosi nella cellula ospite.
L' agente patogeno aumenta o impedisce la sua internalizzazione inducendo o inibendo la fagocitosi della cellula ospite . Vibrio parahaemolyticus è un batterio Gram-negativo che causa intossicazione alimentare a causa del consumo di pesce crudo o poco cotto nell'uomo. VopS, un effettore di tipo III trovato in Vibrio parahaemolyticus , contiene un dominio Fic che ha un motivo HPFx(D/E)GN(G/K)R conservato che contiene un residuo di istidina essenziale per l'AMPilazione. VopS blocca l'assemblaggio dell'actina modificando il residuo di treonina nella regione dell'interruttore 1 delle Rho GTPasi. Il trasferimento di una frazione AMP utilizzando l'ATP al residuo di treonina determina un ostacolo sterico e quindi impedisce alle Rho GTPasi di interagire con gli effettori a valle. VopS adenila anche RhoA e il ciclo di divisione cellulare 42 (CDC42), portando a una disaggregazione della rete di filamenti di actina. Di conseguenza, il controllo del citoscheletro di actina della cellula ospite è disabilitato, portando all'arrotondamento delle cellule.
L'IbpA è secreto nelle cellule eucariotiche da H. somni , un batterio Gram-negativo nei bovini che causa l'infezione dell'epitelio respiratorio. Questo effettore contiene due domini Fic nella regione C-terminale. L'AMPilazione del dominio IbpA Fic delle GTPasi della famiglia Rho è responsabile della sua citotossicità. Entrambi i domini Fic hanno effetti simili sul citoscheletro delle cellule ospiti come VopS. L'AMPilazione su un residuo di tirosina della regione switch 1 blocca l'interazione delle GTPasi con substrati a valle come PAK.
DrrA è il substrato del sistema di traslocazione di tipo IV Dot/Icm DrrA da Legionella pneumophila . È l'effettore secreto da L. pneumophila per modificare le GTPasi delle cellule ospiti. Questa modifica aumenta la sopravvivenza dei batteri nelle cellule ospiti. DrrA è composto da un dominio specifico del fattore di scambio dei nucleotidi guanina (GEF) Rab1b , un dominio di legame lipidico C-terminale e un dominio N-terminale con proprietà citotossiche poco chiare. I lavori di ricerca mostrano che la DrrA N-terminale e a lunghezza intera mostra l'attività degli AMPilatori verso la proteina Rab1b dell'ospite (proteina correlata a Ras), che è anche il substrato del dominio Rab1b GEF. La proteina Rab1b è la GTPasi Rab per regolare il trasporto delle vescicole e la fusione della membrana. L'adenilazione da parte dei batteri AMPilatori prolunga lo stato di Rab1b legato al GTP. Pertanto, il ruolo dell'effettore DrrA è collegato ai benefici dei vacuoli dei batteri per la loro replicazione durante l'infezione.
eucariotico
Piante e lieviti non hanno enzimi AMPilanti endogeni noti, ma i genomi animali sono dotati di una singola copia di un gene che codifica per un'AMPilasi di dominio Fic, che è stato probabilmente acquisito da un antenato degli animali tramite trasferimento genico orizzontale da un procariota. La proteina umana comunemente indicata come FICD, era stata precedentemente identificata come proteina E associata all'huntingtina (HypE; un'assegnazione derivante da uno schermo a due ibridi di lievito, ma di dubbia rilevanza, poiché Huntingtina e HypE/FICD sono localizzate in diversi compartimenti cellulari) . CG9523 Anche gli omologhi in Drosophila melanogaster (CG9523) e C. elegans (Fic-1) hanno ricevuto attenzione. In tutti gli animali FICD ha una struttura simile. È una proteina del dominio transmembrana di tipo II , con un breve dominio citoplasmatico seguito da un ancoraggio membranaceo che trattiene la proteina nel reticolo endoplasmatico (ER) e una lunga porzione C-terminale che risiede nell'ER e comprende ripetizioni tetratricopeptidiche (TPR) seguite da un catalizzatore dominio Fic.
Reticolo endoplasmatico
La scoperta di una cellula animale AMPylase, seguita dalla scoperta della sua localizzazione ER e che BiP è un substrato importante per la sua attività, sono state importanti scoperte. BiP (noto anche come Grp78) era noto da tempo per subire una modifica post-traduzionale inattivante, ma la sua natura rimane sfuggente. Ampiamente ipotizzato essere ADP-ribosilazione , risulta essere AMPilazione mediata da FICD, poiché l'inattivazione del gene FICD nelle cellule ha abolito tutte le modificazioni post-traduzionali misurabili di BiP.
BiP è un chaperone proteico localizzato nell'ER la cui attività è strettamente regolata a livello trascrizionale tramite un programma di espressione genica noto come Unfolded Protein Response (UPR). L'UPR è un processo omeostatico che accoppia il tasso di trascrizione di BiP (e di molte altre proteine) al carico delle proteine non ripiegate nell'ER (il cosiddetto stress ER) per aiutare a mantenere la proteostasi dell'ER . L'AMPilazione aggiunge un altro rapido livello post-traduzionale di controllo dell'attività di BiP, poiché la modifica di Thr518 del dominio di legame del substrato di BiP con un AMP blocca il chaperone in una conformazione inattiva. Questa modifica viene implementata selettivamente quando lo stress ER diminuisce, per inattivare il BiP in eccesso. Tuttavia, quando lo stress ER aumenta di nuovo, lo stesso enzima, FICD, catalizza la reazione opposta, la de-AMPilazione di BiP.
Un undertanding della base strutturale di BiP AMPylation e de-AMPylation sta gradualmente emergendo, come sono indizi per l'allosteria che possa regolare l'interruttore Attività FICD ma importanti dettagli di questo processo si verifica in cellule rimangono da scoprire.
Il ruolo di FICD nell'AMPilazione (e de-AMPilazione) di BiP su Thr518 è ben supportato da studi biochimici e strutturali. È stata anche presentata la prova che in alcune circostanze FICD può AMPilare un residuo diverso, Thr366 nel dominio di legame del nucleotide di BiP.
Caenorhabditis elegans
Fic-1 è l'unica proteina Fic presente nel codice genetico di C. elegans . Si trova principalmente nell'involucro nucleare ER delle cellule germinali adulte e delle cellule embrionali, ma piccole quantità possono essere trovate all'interno del citoplasma. Questo pool extra-ER di FICD-1 è accreditato con l'AMPilazione degli istoni del nucleo e fattori di traduzione di tipo eEF1-A all'interno del nematode.
Sebbene i diversi livelli di AMPilazione non abbiano creato effetti evidenti all'interno del comportamento o della fisiologia del nematode, i vermi knockout Fic-1 erano più suscettibili all'infezione da Pseudomonas aeruginosa rispetto alle controparti con domini Fic-1 attivi, il che implica un legame tra AMPilazione di bersagli cellulari e risposte immunitarie all'interno dei nematodi.
Drosophila melanogaster
Le mosche prive di FICD (CG9523) sono state descritte come cieche. Inizialmente, questo difetto è stato attribuito a un ruolo della FICD sulla superficie cellulare delle proiezioni capitate - un sito putativo di riciclo dei neurotrasmettitori, tuttavia uno studio successivo ha implicato l'AMPilazione mediata da FICD di BiP Thr366 nel problema visivo
significato clinico
La proteina presinaptica α-sinucleina è risultata essere un bersaglio per l'AMPilazione FICD. Durante l'adenilazione di αSyn mediata da HypE , l'aggregazione di αSyn diminuisce e si è scoperto che sia la neurotossicità che lo stress ER diminuiscono in vitro . Pertanto, l'adenilazione di αSyn è probabilmente una risposta protettiva allo stress ER e all'aggregazione di αSyn. Tuttavia, poiché aSyn e FICD risiedono in compartimenti diversi, è necessario effettuare ulteriori ricerche per confermare l'importanza di queste affermazioni.
rilevamento
Maniglie chimiche
Le maniglie chimiche vengono utilizzate per rilevare le proteine modificate post-traduzionali. Recentemente, c'è un N6pATP che contiene un tag alchinile (propargil) nella posizione N6 dell'adenina dell'ATP. Questo N6pATP si combina con la reazione click per rilevare le proteine AMPilate. Per rilevare proteine modificate non riconosciute ed etichettare substrati VopS, vengono utilizzati derivati dell'ATP con un fluoroforo sull'adenina N6 NH2.
Metodo basato su anticorpi
L'anticorpo è famoso per la sua elevata affinità e selettività, quindi è il modo migliore per rilevare le proteine AMPilate. Recentemente, gli anticorpi ɑ-AMP vengono utilizzati per rilevare e isolare direttamente le proteine AMPilate (in particolare la tirosina AMPilata e la treonina AMPilata) dalle cellule e dai lisati cellulari. L'AMPilazione è una modifica post-traduzionale, quindi modificherà le proprietà delle proteine conferendo il carattere polare dell'AMP e l'idrofobicità. Pertanto, invece di utilizzare anticorpi che rilevano un'intera sequenza peptidica, è preferibile aumentare gli anticorpi AMP direttamente mirati a specifici amminoacidi.
Spettrometria di massa
In precedenza, molti lavori scientifici utilizzavano la spettrometria di massa (MS) in diverse modalità di frammentazione per rilevare i peptidi AMPilati. In risposta alle tecniche di frammentazione distintive, le sequenze proteiche AMPilate si sono disintegrate in diverse parti dell'AMP. Mentre la dissociazione per trasferimento di elettroni (ETD) crea frammenti minimi e spettri meno complicati, la dissociazione indotta da collisione (CID) e la frammentazione da collisione ad alta energia (HCD) generano ioni caratteristici adatti all'identificazione di proteine AMPilate generando più frammenti di AMP. Grazie alla stabilità dell'AMP, gli spettri di frammentazione peptidica sono facili da leggere manualmente o con i motori di ricerca.
Inibitori
Sono stati scoperti inibitori dell'AMPilazione proteica con costante inibitoria (K i ) compresa tra 6 e 50 µM e selettività almeno 30 volte rispetto a HypE.