Alfavirus -Alphavirus
e
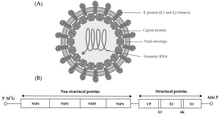
Alphavirus è un genere di virus a RNA , l'unico genere dellafamiglia Togaviridae . Gli alfavirus appartengono al gruppo IV della classificazione dei virus di Baltimora , con ungenoma a RNA a singolo filamento a senso positivo . Ci sono 32 alfavirus, che infettano vari vertebrati come umani, roditori, pesci, uccelli e mammiferi più grandi come i cavalli, così come gli invertebrati . Gli alfavirus che potrebbero infettare sia i vertebrati che gli artropodi sono indicati come alfavirus a doppio ospite, mentre gli alfavirus specifici per gli insetti come il virus Eilat e il virus Yada yada sono limitati al loro vettore artropodi competente. La trasmissione tra specie e individui avviene principalmente attraverso le zanzare, rendendo gli alphavirus un membro della collezione di arbovirus – ovirus trasmessi da artropodi . Le particelle di alfavirus sono avvolte, hanno un diametro di 70 nm, tendono ad essere sferiche (anche se leggermente pleomorfe ) e hanno un nucleocapside isometrico di 40 nm.
genoma
| Alpha_E1_glycop | |||||||||
|---|---|---|---|---|---|---|---|---|---|
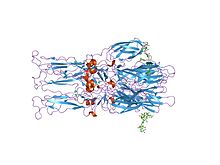 Struttura cristallina del omotrimero della fusione glicoproteina E1 da Semliki Forest Virus
| |||||||||
| Identificatori | |||||||||
| Simbolo | Alpha_E1_glycop | ||||||||
| Pfam | PF01589 | ||||||||
| InterPro | IPR002548 | ||||||||
| SCOP2 | 1rer / SCOPe / SUPFAM | ||||||||
| TCDB | 1.G | ||||||||
| Superfamiglia OPM | 109 | ||||||||
| proteine OPM | 1rer | ||||||||
| |||||||||
| Alpha_E2_glycop | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 Mappatura della glicoproteina E2 degli alfavirus
| |||||||||
| Identificatori | |||||||||
| Simbolo | Alpha_E2_glycop | ||||||||
| Pfam | PF00943 | ||||||||
| InterPro | IPR000936 | ||||||||
| TCDB | 1.G | ||||||||
| Superfamiglia OPM | 109 | ||||||||
| proteine OPM | 2tasso | ||||||||
| |||||||||
| Alpha_E3_glycop | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Identificatori | |||||||||
| Simbolo | Alpha_E3_glycop | ||||||||
| Pfam | PF01563 | ||||||||
| InterPro | IPR002533 | ||||||||
| TCDB | 1.G | ||||||||
| Superfamiglia OPM | 109 | ||||||||
| |||||||||
Gli alfavirus sono virus piccoli, sferici, avvolti con un genoma di un singolo filamento di RNA di senso positivo. La lunghezza totale del genoma varia tra 11.000 e 12.000 nucleotidi e ha un cappuccio 5' e una coda 3' poli-A . I quattro geni proteici non strutturali sono codificati nei due terzi 5' del genoma, mentre le tre proteine strutturali sono tradotte da un mRNA subgenomico colineare con il terzo 3' del genoma.
Ci sono due strutture di lettura aperte (ORF) nel genoma, non strutturali e strutturali. Il primo è non strutturale e codifica per proteine (nsP1-nsP4) necessarie per la trascrizione e la replicazione dell'RNA virale. Il secondo codifica tre proteine strutturali : la proteina nucleocapside C del nucleo e le proteine dell'involucro P62 ed E1, che si associano come eterodimero . Le glicoproteine di superficie ancorate alla membrana virale sono responsabili del riconoscimento del recettore e dell'ingresso nelle cellule bersaglio attraverso la fusione della membrana .
proteine strutturali
La maturazione proteolitica di P62 in E2 ed E3 provoca un cambiamento nella superficie virale. Insieme, i "picchi" di glicoproteina E1, E2 e talvolta E3, formano un dimero E1/E2 o un trimero E1/E2/E3, dove E2 si estende dal centro ai vertici, E1 riempie lo spazio tra i vertici ed E3, se presente, si trova all'estremità distale della punta. Dopo l'esposizione del virus all'acidità dell'endosoma , E1 si dissocia da E2 per formare un omotrimero E1 , necessario per la fase di fusione per guidare insieme le membrane cellulari e virali . La glicoproteina alfavirale E1 è una proteina di fusione virale di classe II, strutturalmente diversa dalle proteine di fusione di classe I che si trovano nel virus dell'influenza e nell'HIV. La struttura del virus della foresta di Semliki ha rivelato una struttura simile a quella della glicoproteina E flavivirale, con tre domini strutturali nella stessa sequenza primaria . La glicoproteina E2 funziona per interagire con il nucleocapside attraverso il suo dominio citoplasmatico , mentre il suo ectodominio è responsabile del legame con un recettore cellulare . La maggior parte degli alfavirus perde la proteina periferica E3, ma nei virus Semliki rimane associata alla superficie virale.
Proteine non strutturali
Quattro proteine non strutturali (nsP1-4) che sono prodotte come una singola poliproteina costituiscono il meccanismo di replicazione del virus. L'elaborazione della poliproteina avviene in modo altamente regolato, con la scissione alla giunzione P2/3 che influenza l'uso del modello di RNA durante la replicazione del genoma. Questo sito si trova alla base di una stretta fessura e non è facilmente accessibile. Una volta scisso nsP3 crea una struttura ad anello che circonda nsP2. Queste due proteine hanno un'interfaccia estesa.
Mutazioni in nsP2 che producono virus non citopatici o un cluster di fenotipi sensibili alla temperatura nella regione dell'interfaccia P2/P3. Le mutazioni di P3 opposte alla posizione delle mutazioni non citopatiche di nsP2 impediscono una scissione efficiente di P2/3. Questo a sua volta influenza l'infettività dell'RNA alterando i livelli di produzione di RNA virale.
Virologia
Il virus ha un diametro di 60-70 nanometri . È avvolto, sferico e ha un genoma di RNA a filamento positivo di ~ 12 kilobasi. Il genoma codifica per due poliproteine. La prima poliproteina consiste di quattro unità non strutturali: nell'ordine dal terminale N al terminale C - nsP1, nsP2, nsP3 e nsP4. La seconda è una poliproteina strutturale composta da cinque unità di espressione: dal terminale N al terminale C - Capside, E3, E2, 6K ed E1. Un RNA a filamento positivo subgenomico - l'RNA 26S - viene replicato da un intermedio di RNA a filamento negativo. Questo serve come modello per la sintesi delle proteine strutturali virali. La maggior parte degli alfavirus ha conservato domini coinvolti nella regolazione della sintesi dell'RNA virale.
Il nucleocapside, di 40 nanometri di diametro, contiene 240 copie della proteina del capside e ha una simmetria icosaedrica T = 4. Le glicoproteine virali E1 ed E2 sono incorporate nel doppio strato lipidico. Singole molecole E1 ed E2 si associano per formare eterodimeri. Gli eterodimeri E1-E2 formano contatti uno a uno tra la proteina E2 e i monomeri nucleocapside. Le proteine E1 ed E2 mediano il contatto tra il virus e la cellula ospite.
Sono stati identificati diversi recettori. Questi includono proibitina , fosfatidilserina , glicosaminoglicani e subunità dell'ATP sintasi .
La replicazione avviene all'interno del citoplasma e i virioni maturano per gemmazione attraverso la membrana plasmatica, dove vengono assimilate le glicoproteine di superficie E2 ed E1 codificate dal virus.
Queste due glicoproteine sono i bersagli di numerose reazioni e test sierologici, tra cui la neutralizzazione e l'inibizione dell'emoagglutinazione. Gli alfavirus mostrano vari gradi di cross-reattività antigenica in queste reazioni e questo costituisce la base per i sette complessi antigenici, 32 specie e molti sottotipi e varietà. La proteina E2 è il sito della maggior parte degli epitopi neutralizzanti, mentre la proteina E1 contiene epitopi cross-reattivi più conservati.
Evoluzione
Uno studio su questo taxon suggerisce che questo gruppo di virus aveva un'origine marina, in particolare l'Oceano Meridionale, e che si sono successivamente diffusi sia nel Vecchio che nel Nuovo Mondo.
Ci sono tre sottogruppi in questo genere: il sottogruppo del virus della foresta di Semliki (virus della foresta di Semliki, O'nyong-nyong e del fiume Ross); il sottogruppo del virus dell'encefalite equina orientale (encefalite equina orientale e virus dell'encefalite equina venezuelana) e il sottogruppo del virus Sindbis. Il virus Sindbis, geograficamente limitato al Vecchio Mondo, è più strettamente correlato al sottogruppo dell'encefalite equina orientale, che sono virus del Nuovo Mondo, rispetto al sottogruppo del virus della foresta di Semliki che si trova anche nel Vecchio Mondo.
Tassonomia
Le seguenti specie sono assegnate al genere:
- Virus dell'aura
- Virus della foresta di Barmah
- virus Bebaru
- virus Caaingua
- virus Cabassou
- Virus Chikungunya
- Virus dell'encefalite equina orientale
- Eilat virus
- Virus delle Everglades
- Virus di Fort Morgan
- virus getah
- Virus delle Highlands J
- virus Madariaga
- Virus Mayaro
- Virus di Middelburg
- Virus Mosso das Pedras
- Virus Mucambo
- virus Ndumu
- O'nyong'nyong virus
- Virus Pixuna
- Virus Rio Negro
- Virus del fiume Ross
- Virus della malattia del pancreas di salmone
- Virus della foresta di Semliki
- Sindbis virus
- Virus dell'elefante marino del sud
- Tonate virus
- virus trocara
- Un virus
- Virus dell'encefalite equina venezuelana
- Virus dell'encefalite equina occidentale
- Virus Whataroa
I sette complessi sono:
- Complesso del virus della foresta di Barmah
-
Complesso di encefalite equina orientale
- Virus dell'encefalite equina orientale (sette tipi antigenici)
- Complesso del virus di Middelburg
- Complesso del virus Ndumu
-
Complesso del virus della foresta di Semliki
- virus Bebaru
- Virus Chikungunya
- virus getah
-
Virus Mayaro
- Sottotipo: Una virus
-
O'nyong'nyong virus
- Sottotipo: virus Igbo-Ora
-
Virus del fiume Ross
- Sottotipo: virus Sagiyama
-
Virus della foresta di Semliki
- Sottotipo: Me Tri virus
-
Complesso di encefalite equina venezuelana
- virus Cabassou
- Virus delle Everglades
- Virus Mosso das Pedras
- Virus Mucambo
- virus paramana
- Virus Pixuna
- Virus Rio Negro
- virus trocara
- Sottotipo: virus Bijou Bridge
- Virus dell'encefalite equina venezuelana
- Complesso di encefalite equina occidentale
- Ricombinanti all'interno di questo complesso
- Virus Buggy Creek
- Virus di Fort Morgan
- Virus delle Highlands J
- Virus dell'encefalite equina occidentale
-
Non classificato
- Eilat virus
- Mwinilunga alfavirus
- Alfavirus dei salmonidi
- Virus dell'elefante marino del sud
- Tonate virus
- virus Caaingua
Appunti
Il virus della foresta di Barmah è correlato al virus della foresta di Semliki. Il virus di Middelburg, sebbene classificato come un complesso separato, potrebbe essere un membro del gruppo dei virus della foresta di Semliki.
Sembra probabile che il genere si sia evoluto nel Vecchio Mondo da un virus vegetale trasmesso da insetti.
Il virus Sindbis potrebbe aver avuto origine in Sud America. I virus dell'encefalite equina e il virus Sindbis sono correlati.
I virus del Vecchio Mondo e del Nuovo Mondo sembrano essersi separati tra 2000 e 3000 anni fa. La divergenza tra il virus dell'encefalite equina venezuelana e il virus dell'equino orientale sembra essere stata circa 1400 anni fa.
Il clade che infetta il pesce sembra essere basale per le altre specie.
Il virus dell'elefante marino del sud sembra essere correlato al clade Sinbis.
Patogenesi e risposta immunitaria
| Virus | Malattia umana | Serbatoio di vertebrati | Distribuzione |
|---|---|---|---|
| Virus della foresta di Barmah | Umani | Australia | |
| Virus Chikungunya | Eruzione cutanea, artrite | Primati, umani | Africa, America Latina, India , Sud-est asiatico |
| Virus dell'encefalite equina orientale | Encefalite | Uccelli | Americhe |
| Virus Mayaro | Eruzione cutanea, artrite | Primati, umani | Sud America |
| O'nyong'nyong virus | Eruzione cutanea, artrite | Primati , Umani | Africa |
| Virus del fiume Ross | Eruzione cutanea, artrite | Mammiferi, umani | Australia, Sud Pacifico |
| Virus della foresta di Semliki | Eruzione cutanea, artrite | Uccelli | Africa |
| Sindbis virus | Eruzione cutanea, artrite | Uccelli | Europa , Africa , Australia |
| Tonate virus | Encefalite | Umani | Sud America |
| Un virus | Eruzione cutanea, artrite | Primati, umani | Sud America |
| Virus dell'encefalite equina venezuelana | Encefalite | Roditori , cavalli | Americhe |
| Virus dell'encefalite equina occidentale | Encefalite | Uccelli, mammiferi | Nord America |
Ci sono molti alphavirus distribuiti in tutto il mondo con la capacità di causare malattie umane. Artrite infettiva , encefalite , eruzioni cutanee e febbre sono i sintomi più comunemente osservati. Mammiferi più grandi come umani e cavalli sono di solito ospiti senza uscita o svolgono un ruolo minore nella trasmissione virale; tuttavia, nel caso dell'encefalite equina venezuelana, il virus è principalmente amplificato nei cavalli. Nella maggior parte degli altri casi il virus si mantiene in natura nelle zanzare, nei roditori e negli uccelli.
Le infezioni da alfavirus terrestri sono trasmesse da insetti vettori come le zanzare. Una volta che un essere umano viene morso dalla zanzara infetta, il virus può entrare nel flusso sanguigno, causando la viremia . L'alphavirus può anche entrare nel SNC dove è in grado di crescere e moltiplicarsi all'interno dei neuroni. Questo può portare a encefalite , che può essere fatale.
Quando un individuo viene infettato da questo particolare virus, il suo sistema immunitario può svolgere un ruolo nell'eliminazione delle particelle virali. Gli alfavirus sono in grado di provocare la produzione di interferoni . Sono coinvolti anche anticorpi e cellule T. Gli anticorpi neutralizzanti svolgono anche un ruolo importante per prevenire ulteriori infezioni e diffusione.
Diagnosi, prevenzione e controllo
La diagnosi si basa su campioni clinici dai quali il virus può essere facilmente isolato e identificato. Non sono attualmente disponibili vaccini contro l'alfavirus. Il controllo dei vettori con repellenti, indumenti protettivi, distruzione dei siti di riproduzione e irrorazione sono le misure preventive di scelta.
Ricerca
Gli alfavirus sono di interesse per i ricercatori di terapia genica , in particolare il virus Ross River, il virus Sindbis , il virus della foresta di Semliki e il virus dell'encefalite equina venezuelana sono stati tutti utilizzati per sviluppare vettori virali per la consegna genica. Di particolare interesse sono i virus chimerici che possono formarsi con involucri alfavirali e capsidi retrovirali. Tali chimere sono chiamate virus pseudotipizzati. Gli pseudotipi dell'involucro alfavirale di retrovirus o lentivirus sono in grado di integrare i geni che trasportano nell'ampia gamma di potenziali cellule ospiti che sono riconosciute e infettate dalle proteine dell'involucro alfavirale E2 ed E1. L'integrazione stabile dei geni virali è mediata dagli interni retrovirali di questi vettori. Ci sono limitazioni all'uso di alfavirus nel campo della terapia genica a causa della loro mancanza di targeting, tuttavia, attraverso l'introduzione di domini anticorpali variabili in un loop non conservato nella struttura di E2, sono state prese di mira specifiche popolazioni di cellule. Inoltre, l'uso di alfavirus interi per la terapia genica è di limitata efficacia sia perché diverse proteine alfavirali interne sono coinvolte nell'induzione dell'apoptosi dopo l'infezione, sia perché il capside alfavirale media solo l'introduzione transitoria di mRNA nelle cellule ospiti. Nessuna di queste limitazioni si estende agli pseudotipi dell'involucro alfavirale di retrovirus o lentivirus. Tuttavia, l'espressione degli involucri del virus Sindbis può portare all'apoptosi e la loro introduzione nelle cellule ospiti dopo l'infezione da parte dei retrovirus pseudotipizzati dell'involucro del virus Sindbis può anche portare alla morte cellulare. La tossicità degli involucri virali Sindbis può essere la causa dei titoli di produzione molto bassi realizzati da cellule di confezionamento costruite per produrre pseudotipi Sindbis. Un altro ramo di ricerca che coinvolge gli alfavirus è la vaccinazione. Gli alfavirus possono essere progettati per creare vettori di repliconi che inducono efficacemente risposte immunitarie umorali e delle cellule T. Potrebbero quindi essere usati per vaccinare contro antigeni virali, batterici, protozoari e tumorali.
Storia
Inizialmente, la famiglia Togaviridae includeva quelli che oggi sono chiamati Flavivirus , all'interno del genere Alphavirus . I flavivirus si sono formati nella propria famiglia quando sono state notate differenze sufficienti con gli alfavirus a causa dello sviluppo del sequenziamento. Il virus della rosolia era precedentemente incluso nella famiglia Togaviridae nel proprio genere Rubivirus , ma ora è classificato nella propria famiglia Matonaviridae . Alphavirus è ora l'unico genere della famiglia.
- 1930 – Il virus dell'encefalite equina occidentale viene isolato per la prima volta negli Stati Uniti (il primo alfavirus mai isolato)
- 1933 – Il virus dell'encefalite equina orientale viene isolato per la prima volta negli Stati Uniti.
- 1938 – Viene isolata l'encefalite equina venezuelana .
- 1941 – Negli Stati Uniti si osserva un'epidemia di encefalite equina occidentale . Colpisce 300.000 cavalli e 3.336 umani.
- 1941 – Norman Gregg nota un gran numero di bambini affetti da cataratta a seguito di un'epidemia di rosolia. Questo e altri difetti vengono quindi classificati sotto la sindrome della rosolia congenita .
- 1942 – Il virus della foresta di Semliki viene isolato a Buliyama, nella contea di Bwamba, in Uganda .
- 1952 – Il virus Sindbis viene isolato nel distretto sanitario di Sindbis, 40 miglia (64 km) a nord del Cairo, in Egitto .
- 1959 – Il virus del fiume Ross viene isolato dalle zanzare Aedes vigilax (ora conosciute come Ochlerotatus vigilax ) che sono state intrappolate nel fiume Ross in Australia.
- 1963 – Il virus del fiume Ross, che causa la poliartrite epidemica (soprattutto osservata in Australia), viene isolato da Doherty e colleghi.
- 1971 – Ultima epidemia di encefalite equina venezuelana nei cavalli nel sud del Texas .
- 1986 – Il virus della foresta di Barmah viene identificato come causa di malattie umane in Australia.
- 2001 – Gli scienziati hanno risolto la struttura cristallina del guscio glicoproteico del virus della foresta di Semliki .
- 2005-2006 - Grande epidemia del virus chikungunya nell'isola di La Réunion e nelle isole circostanti nell'Oceano Indiano
- 2006 – Grande epidemia del virus chikungunya in India con oltre 1,5 milioni di casi segnalati
Guarda anche
Fonti
- "Arbovirus" . Virologia-online .
- "Fonti ICTV" . ICTV . Archiviato dall'originale il 12 febbraio 2006.
- Smerdou, C.; Liljestrom, P. (2000). "Vettori Alphavirus: dalla produzione di proteine alla terapia genica". Terapia genica e regolazione . 1 (1): 33-63. ISSN 1568-5586 .
- Rayner JO, Dryga SA, Kamrud KI (2002). "Vettori alfavirus e vaccinazione". Recensioni in Virologia Medica . 12 (5): 279-96. doi : 10.1002/rmv.360 . PMID 12211042 . S2CID 21432844 .
- Rhême, Céline; Ehrengruber, Markus U.; Grandgirard, Denis. "Citotossicità alfavirale e sue implicazioni nello sviluppo del vettore" . Fisiologia Sperimentale . 90 (1): 45–52. doi : 10.1113/expphysiol.2004.028142 . PMID 15542620 .
- Schmaljohn, Alan L.; McClain, David (1996). "54. Alfavirus (Togaviridae) e Flavivirus (Flaviviridae)" . In Baron, Samuele (ed.). Microbiologia medica (4a ed.). Filiale medica dell'Università del Texas a Galveston. ISBN 0-9631172-1-1. PMID 21413253 . NBK7633.