DMSO reduttasi - DMSO reductase
| Dimetilsolfossido reduttasi | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Identificatori | |||||||||
| Numero CE | 1.8.5.3 | ||||||||
| Banche dati | |||||||||
| IntEnz | Vista IntEnz | ||||||||
| BRENDA | Entrata BRENDA | ||||||||
| ExPASy | Vista NiceZyme | ||||||||
| KEGG | Ingresso KEGG | ||||||||
| MetaCyc | passaggio metabolico | ||||||||
| PRIAM | profilo | ||||||||
| Strutture PDB | RCSB PDB PDBe PDBsum | ||||||||
| |||||||||
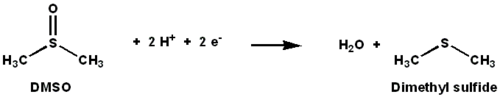
La DMSO reduttasi è un enzima contenente molibdeno che catalizza la riduzione del dimetilsolfossido (DMSO) a dimetil solfuro (DMS). Questo enzima funge da reduttasi terminale in condizioni anaerobiche in alcuni batteri, con DMSO che è l'accettore di elettroni terminale. Durante il corso della reazione, l'atomo di ossigeno in DMSO viene trasferito in molibdeno e quindi ridotto in acqua.
La DMSO reduttasi (DMSOR) e altri membri della famiglia DMSO reduttasi sono unici per batteri e archeobatteri . Enzimi di questa famiglia nella fosforilazione ossidativa anaerobica e nella respirazione litotrofica basata su donatori inorganici . Questi enzimi sono stati progettati per degradare gli oxoanioni. DMSOR catalizza il trasferimento di due elettroni e un atomo di ossigeno nella reazione: il sito attivo di DMSOR contiene molibdeno, altrimenti raro in biologia.
Struttura terziaria e sito attivo
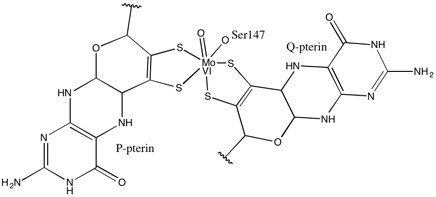
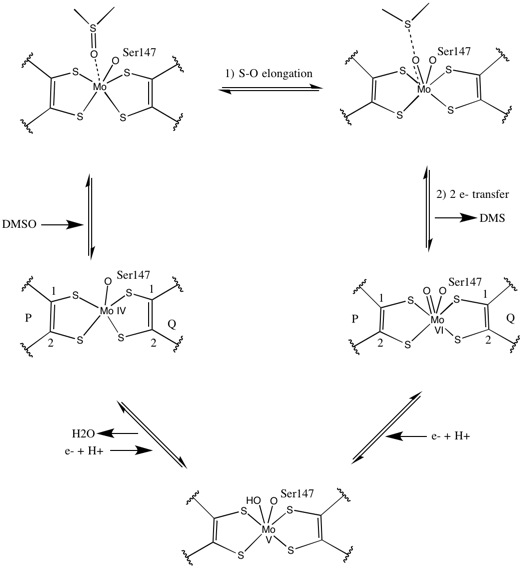
Come per altri membri della famiglia DMSO reduttasi, la struttura terziaria di DMSOR è composta da domini Mo-circostanti I-IV, con dominio IV fortemente interagente con piranopterinditiolene Mo-cofattori (P- e Q-pterina) del sito attivo. I membri della famiglia DMSO reduttasi differiscono in termini di siti attivi. Nel caso di DMSOR, il centro Mo si trova a due ditiolene fornito da due cofattori piranotteri. Questi cofattori organici, chiamati molibdopterine , sono collegati a GMP per creare una forma dinucleotidica. Un ulteriore quinto ligando simile a un cappuccio è la catena laterale O del residuo di serina-147, che classifica ulteriormente l'enzima come riduttasi DMSO di tipo III. InTipo I e II la serina è sostituita rispettivamente da residui di cisteina e aspartato. A seconda dello stato redox del Mo, che oscilla tra IV, V o VI man mano che la reazione progredisce, il nucleo del sito attivo Mo può anche essere legato a un atomo di ossigeno di un gruppo acqua, idrosso o osso, rispettivamente . Gli studi hanno dimostrato che la particolare identità dell'amminoacido utilizzato per coordinare il nucleo del Mo influenza notevolmente il potenziale del punto medio redox del Mo e lo stato di protonazione della legatura del gruppo dell'ossigeno, che sono determinanti chiave nel meccanismo di catalisi dell'enzima.
Meccanismo
Gli studi iniziali di DMSO 18 isotopici hanno stabilito un meccanismo a doppia ossotransferasi per DMSOR di R. sphaeroides . In questo meccanismo O etichetta 18 viene trasferito dal substrato Mo, che poi trasferisce la O 18 di 1,3,5-triaza-7-phosphaadamantane (PTA) per ottenere PTAO 18 . In un meccanismo analogo, DMSO trasferisce O a Mo e il centro Mo (VI) O risultante viene ridotto, producendo acqua.
Studi sui complessi sintetici di Mo bis- ditiolene suggeriscono che si tratti di trasferimento di ossigeno, trasferimento di elettroni. Utilizzando S K-edge XAS e DFT, questi studi modello puntano a scissione SO concordata e trasferimento di elettroni. I tassi sono proporzionali alla diminuzione della forza di legame XO del substrato e all'aumento dell'affinità protonica del substrato.
La cristallografia a raggi X ha stabilito che la struttura terziaria complessiva dell'enzima rimane costante durante la progressione della reazione. Tuttavia, diversi diversi esperimenti condotti su DMSOR di R. sphaeroides hanno riportato risultati diversi per l'attività di coordinazione dei quattro potenziali ligandi del ditiolene. Mentre un'indagine di cristallografia a raggi X ha concluso la coordinazione equidistante di tutti e quattro i ligandi Mo-S nella forma ossidata, che è supportata da numerosi studi di spettroscopia di assorbimento di raggi X (XAS), uno studio diverso ha caratterizzato distanze Mo-S asimmetriche. Entrambi gli studi e gli studi di risonanza paramagnetica elettronica (EPR) hanno previsto che il sito attivo Mo è altamente flessibile in termini di posizione e grado di potenziali coordinazioni del ligando.
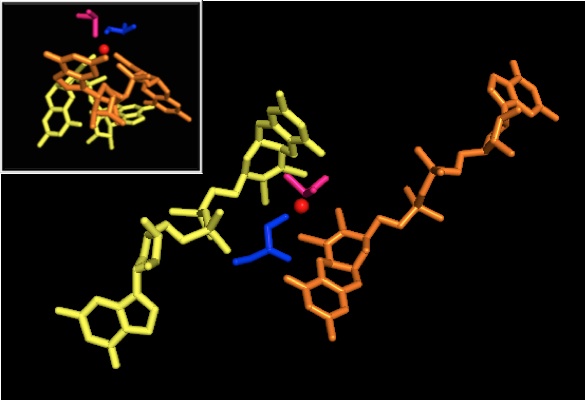
I dati che suggerivano due cofattori di piranopterina significativamente asimmetrici sono stati utilizzati per proporre un meccanismo di reazione. Nella forma Mo VI completamente ossidata del sito attivo, i ligandi del gruppo oxo e della serina sono stati coordinati a una distanza di 1,7 A dal centro Mo. S1 e S2 della P-pterina e S1 della Q-pterina erano posizionate a 2,4 A di distanza dal Mo, e S2 di Q-pterina si trovava a 3,1 A di distanza. Questa asimmetria della pterina può essere il risultato dell'effetto trans del gruppo oxo che indebolisce il legame S2-Mo, che si trova direttamente di fronte al gruppo oxo.
Al contrario, la struttura della forma Mo IV completamente ridotta del sito attivo mostrava che S1 e S2 P-pterina e S1 Q-pterina mantenevano la piena coordinazione, tuttavia l'S2 della Q-pterina si allontanava dal centro metallico, indicando una diminuzione della coordinazione . Questo spostamento nella lunghezza del legame ligando-Mo è coerente con il meccanismo proposto di trasferimento diretto dell'ossigeno dal substrato DMSO al Mo. Una coordinazione più debole del ditiolene nella forma enzimatica ridotta potrebbe facilitare il legame diretto del S = O. Nella riduzione del Mo e nella protonazione del gruppo oxo, si propone che una sorgente di elettroni citocromo possa legarsi a una depressione sopra il sito attivo e ridurre direttamente il centro Mo, o in alternativa questo citocromo potrebbe legarsi a un loop polipeptidico ben solvatato in prossimità della Q-pterina e la Q-pterina potrebbe mediare questo trasferimento di elettroni.
Localizzazione e regolazione cellulare
In R. sphaeroides , DMSOR è una proteina idrosolubile a subunità singola che non richiede cofattori aggiuntivi oltre alla pterina. In E. coli , DMSOR è incorporato all'interno della membrana e ha tre subunità uniche, una delle quali include il caratteristico cofattore pterina, un'altra che contiene quattro cluster 4Fe: 4S e una subunità transmembrana finale che lega e ossida il menachinolo. Il trasferimento di un e- dal menachinolo ai cluster 4Fe: 4S e infine al sito attivo di pterina-Mo genera un gradiente protonico utilizzato per la generazione di ATP.
DMSOR regolato prevalentemente a livello trascrizionale. È codificato dal gene dor ed è espresso quando attivato da una cascata di segnali, che è regolata dalle proteine DorS, DorR e DorC. Uno studio sulle fusioni lacZ (geni reporter) ai corrispondenti promotori dorS, dorR e dorC ha concluso che l'espressione di DorR e DorC aumenta in ambienti con ossigeno ridotto, ma l'espressione di DorS non è stata influenzata dalla concentrazione di ossigeno. L'espressione di DorC è aumentata anche con l'aumento delle concentrazioni di DMSO.
Impatto ambientale
Il DMS, un prodotto di DMSOR, è un componente del ciclo dello zolfo . Il DMS viene ossidato a metansolfonati , che nucleari la condensazione delle nuvole sugli oceani aperti, dove la fonte alternativa di nucleazione, la polvere, è assente. La formazione delle nuvole è una componente chiave per aumentare l'albedo terrestre e regolare la temperatura atmosferica, quindi questo enzima e la reazione che catalizza potrebbero rivelarsi utili sulla frontiera del controllo del clima.