Genetica e archeogenetica dell'Asia meridionale - Genetics and archaeogenetics of South Asia
Genetica e archeogenetica dell'Asia del Sud è lo studio delle genetica e archeogenetica dei gruppi etnici dell'Asia meridionale . Mira a scoprire la storia genetica di questi gruppi . La posizione geografica dell'Asia meridionale rende la sua biodiversità importante per lo studio della dispersione precoce degli esseri umani anatomicamente moderni in tutta l' Asia .
Gli studi basati sulle variazioni del DNA mitocondriale ( mtDNA ) hanno riportato l'unità genetica in varie sottopopolazioni dell'Asia meridionale, mostrando che la maggior parte dei nodi ancestrali dell'albero filogenetico di tutti i tipi di mtDNA ha avuto origine nell'Asia meridionale. Le conclusioni degli studi basati sulla variazione del cromosoma Y e sulla variazione del DNA autosomico sono state variate.
Recenti studi sul genoma mostrano che i sud-asiatici sono discendenti di una componente indigena dell'Asia meridionale (denominata antichi indiani ancestrali del sud , "AASI", lontanamente imparentati con gli andamanesi , i papuani e i nativi australiani), e successivamente arrivati dall'Eurasia occidentale (europei/medi componenti dell'Est) e dell'Eurasia orientale (relativi all'Asia orientale) rispettivamente in gradi diversi.
Gli studi genomici hanno descritto il paesaggio genetico dell'Asia meridionale come un composto di componenti esogeni dell'Eurasia occidentale e dell'Eurasia orientale che si sono mescolati con i gruppi indigeni dell'Asia meridionale per creare i moderni asiatici del sud. La componente di discendenza correlata all'Asia orientale sta formando la maggiore discendenza tra le popolazioni specifiche ai piedi dell'Himalaya e dell'India nordorientale , ed è generalmente distribuita in tutto il subcontinente indiano , con un picco tra i gruppi di lingua austroasiatica , nonché tra i singalesi e i bengalesi .
È stato scoperto che il nodo ancestrale dell'albero filogenetico di tutti i tipi di mtDNA ( aplogruppi del DNA mitocondriale ) tipicamente presenti in Asia centrale, Asia occidentale ed Europa si trovano anche in Asia meridionale a frequenze relativamente elevate. Si stima che la divergenza dedotta di questo nodo ancestrale comune si sia verificata poco meno di 50.000 anni fa, a sostegno di un evento "fuori dall'India" per le popolazioni dell'Eurasia occidentale come gli europei, durante il primo Paleolitico. In India, i principali lignaggi materni sono vari sottocladi M , seguiti dai sottolignaggi R e U. I tempi di coalescenza di questi aplogruppi mitocondriali sono stati approssimati fino ad oggi a 50.000 BP.
I principali lignaggi paterni degli indiani, rappresentati dai cromosomi Y , sono gli aplogruppi R1a1 , R2 , H , L , J2 e O2a . Alcuni ricercatori hanno sostenuto che l'aplogruppo Y-DNA R1a1 (M17) è di origine autoctona dell'Asia meridionale. Tuttavia, anche le proposte per un'origine della steppa dell'Asia centrale/eurasiatica per R1a1 sono abbastanza comuni e supportate da diversi studi più recenti.
Panoramica
Si suggerisce che la popolazione indigena dell'Asia meridionale, a volte definita "AASI", abbia avuto origine da una prima popolazione di dispersione meridionale , che ha anche dato origine ai popoli andamanesi , agli indigeni della Nuova Guinea (Papuani) e agli indigeni australiani . La popolazione indigena dell'Asia meridionale è stata isolata geneticamente da altre popolazioni a partire da circa 45.000 anni aC . L'ascendenza indigena dell'Asia meridionale (AASI) nei moderni sud asiatici è combinata, in vari gradi, con le componenti più recenti dell'Eurasia occidentale e dell'Eurasia orientale del periodo neolitico e dell'età del bronzo . Il popolo tribale Paniya è, accanto al popolo Irula e al popolo Soliga , il miglior proxy per l'ascendenza indigena dell'Asia meridionale.
Si ipotizza che i primi antenati eurasiatici occidentali siano arrivati durante il periodo neolitico o già durante l' Olocene , in tandem con l'arrivo di componenti relativi all'Asia orientale durante il primo periodo neolitico con gruppi austroasiatici e tibeto-birmani rispettivamente del sud-est asiatico e dell'Asia orientale .
mtDNA
Gli aplogruppi del mtDNA più frequenti nell'Asia meridionale sono M , R e U (dove U è un discendente di R). Sostenendo a lungo termine il "modello rivale del cromosoma Y", Stephen Oppenheimer ritiene che sia altamente suggestivo che l'India sia l'origine degli aplogruppi del mtDNA eurasiatico che chiama "Eurasian Eves". Secondo Oppenheimer è altamente probabile che quasi tutte le linee materne umane dell'Asia centrale, del Medio Oriente e dell'Europa discendano da sole quattro linee di mtDNA che hanno avuto origine nell'Asia meridionale 50.000-100.000 anni fa.
Macroaplogruppo M
Il macroaplogruppo M , che è considerato un cluster dei lignaggi materni proto-asiatici, rappresenta oltre il 60% del MtDNA dell'Asia meridionale.
Il macroaplotipo M in India comprende molti sottogruppi che differiscono profondamente da altri sottolineaggi dell'Asia orientale, in particolare le popolazioni mongoloidi. Le profonde radici della filogenesi M accertano chiaramente la reliquia dei lignaggi dell'Asia meridionale rispetto ad altri sottolignaggi M (in Asia orientale e altrove) suggerendo l'origine "in situ" di questi sub-aplogruppi nell'Asia meridionale, molto probabilmente in India. Questi lignaggi radicati non sono specifici della lingua e si estendono a tutti i gruppi linguistici in India.
Praticamente tutti i lignaggi MtDNA M dell'Asia centrale moderna sembrano appartenere all'Eurasia orientale ( mongolo ) piuttosto che ai sottotipi dell'Asia meridionale dell'aplogruppo M, il che indica che nessuna migrazione su larga scala dalle attuali popolazioni di lingua turca dell'Asia centrale si è verificata in India. L'assenza dell'aplogruppo M negli europei, rispetto alla sua frequenza altrettanto elevata tra gli asiatici del sud, gli asiatici orientali e in alcune popolazioni dell'Asia centrale, contrasta con le tendenze eurasiatiche occidentali dei lignaggi paterni dell'Asia meridionale.
La maggior parte dei confini del mtDNA esistenti nell'Asia meridionale e sudoccidentale sono stati probabilmente modellati durante l'insediamento iniziale dell'Eurasia da umani anatomicamente moderni.
| aplogruppo | Sottocladi importanti | Popolazioni |
|---|---|---|
| M2 | M2a, M2b | In tutto il continente con scarsa presenza nel Northwest Peaking in Bangladesh, Andhra Pradesh, Tamil Nadu costiero e Sri Lanka |
| M3 | M3a | Concentrato nell'India nordoccidentale Il più alto tra i parsi di Mumbai |
| M4 | M4a | Picchi in Pakistan, Kashmir e Andhra Pradesh |
| M6 | M6a, M6b | Kashmir e vicino alle coste del Golfo del Bengala, Sri Lanka |
| M18 | In tutta l'Asia meridionale con picchi in Rajasthan e Andhra Pradesh |
|
| M25 | Moderatamente frequente in Kerala e Maharashtra ma piuttosto raro altrove in India |
Macroaplogruppo R
Anche il macroaplogruppo R (una suddivisione molto ampia e antica del macroaplogruppo N ) è ampiamente rappresentato e rappresenta il restante 40% del MtDNA dell'Asia meridionale. Una sua suddivisione molto antica e più importante è l' aplogruppo U che, sebbene presente anche nell'Eurasia occidentale , ha diverse subcladi specifiche dell'Asia meridionale.
I più importanti aplogruppi dell'Asia meridionale all'interno di R:
| aplogruppo | Popolazioni |
|---|---|
| R2 | Ampiamente distribuito in tutto il subcontinente |
| R5 | ampiamente distribuito nella maggior parte dell'India. Picchi nella costa sud-occidentale dell'India |
| R6 | diffuso a bassi tassi in tutta l'India. Picchi tra Tamil e Kashmir |
| W | Trovato negli stati nord-occidentali. Picchi in Gujarat, Punjab e Kashmir, la frequenza è bassa altrove. |
Aplogruppo U
L'aplogruppo U è un sub-aplogruppo del macroaplogruppo R. La distribuzione dell'aplogruppo U è un'immagine speculare di quella dell'aplogruppo M: il primo non è stato finora descritto tra gli asiatici orientali, ma è frequente nelle popolazioni europee così come tra gli asiatici del sud. I lignaggi U dell'Asia meridionale differiscono sostanzialmente da quelli europei e anche la loro coalescenza in un antenato comune risale a circa 50.000 anni.
| aplogruppo | Popolazioni |
|---|---|
| U2* | (un paraaplogruppo ) è scarsamente distribuito specialmente nella metà settentrionale dell'Asia meridionale.
Si trova anche nel sud-ovest dell'Arabia. |
| U2a | mostra una densità relativamente alta in Pakistan e nell'India nordoccidentale, ma anche in Karnataka, dove raggiunge la sua maggiore densità. |
| U2b | ha la più alta concentrazione in Uttar Pradesh ma si trova anche in molti altri luoghi, specialmente in Kerala e Sri Lanka.
Si trova anche in Oman. |
| U2c | è particolarmente importante in Bangladesh e nel Bengala occidentale. |
| U2l | è forse la più importante numericamente tra le subcladi U nell'Asia meridionale, raggiungendo concentrazioni particolarmente elevate (oltre il 10%) in Uttar Pradesh, Sri Lanka, Sindh e parti del Karnataka. Ha anche una certa importanza in Oman. L'aplogruppo U2i del mtDNA è soprannominato "Western Eurasian" in Bamshad et al. studio ma "Eurasiatico orientale (per lo più specifico per l'India)" in Kivisild et al. studio. |
| U7 | questo aplogruppo ha una presenza significativa in Gujarat, Punjab e Pakistan. La possibile patria di questo aplogruppo abbraccia il Gujarat (massima frequenza, 12%) e l'Iran perché da lì la sua frequenza diminuisce vertiginosamente sia verso est che verso ovest. |
cromosoma Y
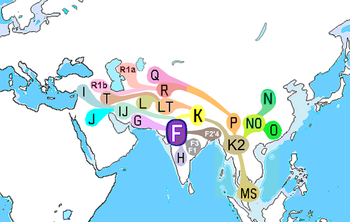
I principali aplogruppi del DNA del cromosoma Y dell'Asia meridionale sono H , J2 , L , R1a1 , R2 e O-K18 . Le loro origini geografiche sono elencate come segue, secondo l'ultima borsa di studio:
| Principali linee cromosomiche Y dell'Asia meridionale: | h | J2 | l | R1a | R2 |
|---|---|---|---|---|---|
| Basu et al. (2003) | nessun commento | nessun commento | nessun commento | Asia centrale | nessun commento |
| Kivisild et al. (2003) | India | Asia occidentale | India | Asia meridionale e occidentale | Asia centro-meridionale |
| Cordaux et al. (2004) | India | Asia occidentale o centrale | Medio orientale | Asia centrale | Asia centro-meridionale |
| Sengupta et al. (2006) | India | Il Medio Oriente e l'Asia centrale | India del sud | India del nord | India del nord |
| Thanseem et al. (2006) | India | Il Levante | Medio Oriente | Asia meridionale e centrale | Asia meridionale e centrale |
| Saho et al. (2006) | Asia del sud | Il Vicino Oriente | Asia del sud | Asia meridionale o occidentale | Asia del sud |
| Mirabal et al. (2009) | nessun commento | nessun commento | nessun commento | India nordoccidentale o Asia centrale | nessun commento |
| Zhao et al. (2009) | India | Medio Oriente | Medio Oriente | Asia centrale o Eurasia occidentale | Asia centrale o Eurasia occidentale |
| Sharma et al. (2009) | nessun commento | nessun commento | nessun commento | Asia del sud | nessun commento |
| Thangaraj et al. (2010) | Asia del sud | Il Vicino Oriente | Il Vicino Oriente | Asia del sud | Asia del sud |
aplogruppo H
L'aplogruppo H (Y-DNA) si trova ad alta frequenza nell'Asia meridionale ed è considerato il principale lignaggio paterno indigeno. H si trova oggi raramente al di fuori dell'Asia meridionale, ma è comune tra le popolazioni discendenti dell'Asia meridionale, come i Romanis , in particolare il sottogruppo H-M82. H è stato trovato anche in alcuni antichi campioni d'Europa e si trova ancora oggi con una bassa frequenza in alcuni europei sudorientali e arabi del Levante . L'aplogruppo H si trova frequentemente tra le popolazioni dell'India , dello Sri Lanka , del Nepal , del Pakistan e delle Maldive . Tutti e tre i rami dell'aplogruppo H (Y-DNA) si trovano nell'Asia meridionale.
Si ritiene che l' aplogruppo H sia sorto nell'Asia meridionale tra 30.000 e 40.000 anni fa. Il suo probabile sito di introduzione è l'Asia meridionale, poiché lì è concentrato. Sembra rappresentare il principale aplogruppo del cromosoma Y degli abitanti paleolitici rispettivamente dell'Asia meridionale e dell'Europa. Alcuni individui dell'Asia meridionale hanno anche dimostrato di appartenere al molto più raro sottoclade H3 (Z5857). L'aplogruppo H non è affatto limitato a popolazioni specifiche. Ad esempio, H è posseduto da circa il 28,8% delle caste indoariane. e nei tribali circa il 25-35%.
Aplogruppo J2
L'aplogruppo J2 è stato presente nell'Asia meridionale principalmente come J2a-M410 e J2b-M102, fin dal neolitico (9500 YBP). I cladi J2 raggiungono le frequenze massime nel nord-ovest e nel sud dell'India e si trovano al 19% nelle caste dell'India meridionale, all'11% nelle caste dell'India settentrionale e al 12% in Pakistan. Nel sud dell'India , la presenza di J2 è più alta tra le caste medie al 21%, seguita dalle caste superiori al 18,6% e dalle caste inferiori al 14%. Tra i gruppi di caste, la più alta frequenza di J2-M172 si osserva tra i Tamil Vellalar dell'India meridionale, al 38,7%. J2 è presente anche nei tribali e ha una frequenza dell'11% nei tribali austro-asiatici. Tra i tribali austro-asiatici, il J2 predominante si trova nel Lodha (35%). J2 è presente anche nel sud dell'India collina tribù Toda al 38.46%, nella tribù Andh di Telangana al 35,19% e nella tribù Kol di Uttar Pradesh ad una frequenza di 33.34%. L'aplogruppo J-P209 è risultato essere più comune nei musulmani sciiti dell'India , di cui il 28,7% appartiene all'aplogruppo J, con il 13,7% in J-M410, il 10,6% in J-M267 e il 4,4% in J2b ( Eaaswarkhanth 2009 ) .
In Pakistan , le frequenze più alte di J2-M172 sono state osservate tra i Parsi al 38,89%, i Brahuis di lingua dravidica al 28,18% e i Makrani Baloch al 24%. Si verifica anche al 18,18% in Makrani Siddis e al 3% in Karnataka Siddis .
J2-M172 si trova con una frequenza complessiva del 10,3% tra il popolo singalese dello Sri Lanka . Nelle Maldive , il 20,6% della popolazione maldiviana è risultato positivo all'aplogruppo J2.
aplogruppo L
Secondo il Dr. Spencer Wells , L-M20 ha avuto origine nella regione del Pamir Knot in Tagikistan ed è migrato in Pakistan e India ca. 30.000 anni fa. Tuttavia, la maggior parte degli altri studi ha proposto un'origine sud-asiatica per L-M20 e ha associato la sua espansione alla valle dell'Indo (~7000 YBP). Ci sono tre sottorami dell'aplogruppo L: L1-M76 (L1a1), L2-M317 (L1b) e L3-M357 (L1a2), che si trovano a vari livelli nell'Asia meridionale.
India
L'aplogruppo L mostra il tempo dell'espansione neolitica. Il clade è presente nella popolazione indiana con una frequenza complessiva di ca. 7-15%. L'aplogruppo L ha una frequenza maggiore tra le caste dell'India meridionale (ca. 17-19%) e raggiunge il 68% in alcune caste del Karnataka, ma è un po' più raro nelle caste dell'India settentrionale (ca. 5-6%). La presenza dell'aplogruppo L è abbastanza rara tra i gruppi tribali (ca. 5,6–7%), tuttavia è stato osservato il 14,6% tra i Chenchus .
Tra i gruppi regionali e sociali, sono state osservate frequenze da moderate ad alte in Konkanastha Brahmins (18,6%), Punjabis (12,1%), Gujaratis (10,4%), Lambadis (17,1%) e Jats (36,8%).
Pakistan
In Pakistan, le sottocladi L1-M76 e L3-M357 di L-M20 raggiungono frequenze complessive rispettivamente del 5,1% e del 6,8%. L'aplogruppo L3 (M357) si trova frequentemente tra Burusho (circa 12%) e Pashtun (circa 7%). La sua più alta frequenza si trova nella provincia sud-occidentale del Balochistan lungo la costa di Makran (28%) fino al delta del fiume Indo . L3a (PK3) si trova in circa il 23% di Nuristani nel nord - ovest del Pakistan .
Il clade è presente in una distribuzione moderata tra la popolazione generale pakistana (14% circa).
Sri Lanka
In uno studio, il 16% dei cingalesi è risultato positivo all'aplogruppo L-M20. In un altro studio il 18% è risultato appartenere a L1.
Aplogruppo R1a1
In Asia meridionale, R1a1 è stato osservato spesso con alta frequenza in un certo numero di gruppi demografici, nonché con la più alta diversità STR che porta alcuni a vederlo come il luogo di origine.
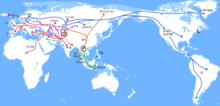
Mentre R1a ha avuto origine ca. Da 22.000 a 25.000 anni fa, la sua sottoclade M417 (R1a1a1) diversificava ca. 5.800 anni fa. La distribuzione delle sottocladi M417 R1-Z282 (incluso R1-Z280) nell'Europa centrale e orientale e R1-Z93 in Asia suggerisce che R1a1a si sia diversificato all'interno delle steppe eurasiatiche o nella regione del Medio Oriente e del Caucaso . Il luogo di origine di queste subcladi gioca un ruolo nel dibattito sulle origini degli indoeuropei . Mentre l'aplogruppo R si trova comunemente tra gli eurasiatici occidentali, studi recenti suggeriscono che il suo lignaggio ancestrale ( aplogruppo P ) discende da una popolazione sud-eurasiatica nel sud-est asiatico . Oggi si trova comunemente tra gli Aeta , i Jahai e alcuni popoli andamanesi .
India
In India , un'alta percentuale di questo aplogruppo si osserva nei bramini del Bengala occidentale (72%) a est, Gujarat Lohanas (60%) a ovest, Khatris (67%) a nord e Iyengar Brahmins (31%) in il Sud. È stato anche trovato in diversi tribali di lingua dravidica dell'India meridionale tra cui i Kota (41%) del Tamil Nadu, i Chenchu (26%) e i Valmiki dell'Andhra Pradesh , nonché gli Yadav e i Kallar del Tamil Nadu, suggerendo che M17 è diffuso in queste tribù indiane del sud. Oltre a questi, gli studi mostrano alte percentuali in gruppi regionali diversi come Manipuris (50%) all'estremo nord-est e tra Punjabis (47%) all'estremo nord-ovest.
Pakistan
In Pakistan si trova al 71% tra i Mohanna della provincia del Sindh a sud e al 46% tra i Balti del Gilgit-Baltistan a nord.
Sri Lanka
Secondo una ricerca del 2003, il 23% dei cingalesi su un campione di 87 soggetti è risultato positivo per R1a1a (R-SRY1532).
Maldive
Alle Maldive , il 23,8% dei maldiviani è risultato positivo per R1a1a (M17).
Nepal
Le persone nella regione di Terai , in Nepal , mostrano R1a1a al 69%.
Aplogruppo R2
In Asia meridionale, la frequenza del lignaggio R2 e R2a è di circa il 10-15% in India e Sri Lanka e del 7-8% in Pakistan. Almeno il 90% degli individui R-M124 si trova nell'Asia meridionale. È segnalato anche nel Caucaso e nell'Asia centrale con una frequenza inferiore. Uno studio genetico di Mondal et al. nel 2017 ha concluso che l' aplogruppo R2 è originario dell'India settentrionale ed era già presente prima della migrazione delle steppe.
India
Tra i gruppi regionali, si trova tra i bengalesi occidentali (23%), gli indù di Nuova Delhi (20%), i punjabi (5%) e i gujarati (3%). Tra i gruppi tribali, Karmalis del Bengala occidentale ha mostrato il più alto al 100% seguito da Lodhas (43%) a est, mentre Bhil del Gujarat a ovest era al 18%, Tharus del nord lo ha mostrato al 17%, Chenchu e Pallan del sud erano rispettivamente al 20% e al 14%. Tra i gruppi di caste, alte percentuali sono mostrate da Jaunpur Kshatriyas (87%), Kamma (73%), Bihar Yadav (50%), Khandayat (46%) e Kallar (44%).
È anche significativamente alto in molti gruppi bramini , tra cui i bramini punjabi (25%), i bramini bengalesi (22%), i bramini Konkanastha (20%), i Chaturvedi (32%), i Bhargava (32%), i pandit del Kashmir (14%) e i Bramini Lingayat (30%).
I musulmani dell'India settentrionale hanno una frequenza del 19% ( sunnita ) e del 13% ( sciiti ), mentre i musulmani Dawoodi Bohra nello stato occidentale del Gujarat hanno una frequenza del 16% e i musulmani Mappila dell'India meridionale hanno una frequenza del 5%.
Pakistan
L'aplogruppo R2 si trova nel 71% della popolazione Burusho . Tra gli Hunza si trova al 18% mentre i Parsi lo mostrano al 20%.
Sri Lanka
Secondo una ricerca del 2003, il 38% dei cingalesi dello Sri Lanka è risultato positivo all'R2.
Maldive
Il 12% dei maldiviani delle Maldive risulta avere R2.
Nepal
In Nepal, le percentuali di R2 variano dal 2% al 26% all'interno di diversi gruppi in vari studi. I Newars mostrano una frequenza significativamente alta del 26% mentre le persone di Kathmandu la mostrano al 10%.
Aplogruppo O2a (O-K18)
In Asia meridionale, la frequenza media di O2a o O-K18 è rispettivamente del 5%-15% in India e del 15%-35% in Sri Lanka e Bangladesh. Tra i gruppi regionali, in particolare tra i gruppi austroasiatici, raggiunge un picco di circa il 75%. L'aplogruppo O2a (O-K18) si trova frequentemente tra gli asiatici del sud-est e dell'est, in particolare tra i coltivatori di riso austroasiatici. Si suggerisce che O2a (O-K18) sia arrivato dal sud-est asiatico in India dopo l'ultimo massimo glaciale, intorno al 10.000 aC.
Ricostruire la storia della popolazione dell'Asia meridionale
L' Indian Genome Variation Consortium (2008) , divide la popolazione dell'Asia meridionale in quattro gruppi etnolinguistici (non genetici): indoeuropeo , dravidico , tibeto-birmano e austro-asiatico . Gli studi di antropologia molecolare utilizzano tre diversi tipi di marcatori: variazione del DNA mitocondriale (mtDNA) che è ereditata dalla madre e altamente polimorfica, variazione del cromosoma Y che coinvolge la trasmissione uniparentale lungo le linee maschili e variazione del DNA autosomico.
variazione del mtDNA
La maggior parte degli studi basati sulla variazione del mtDNA hanno riportato l'unità genetica delle popolazioni dell'Asia meridionale attraverso lingua, casta e gruppi tribali. È probabile che l'aplogruppo M sia stato portato in Asia dall'Africa orientale lungo la rotta meridionale dalla prima ondata migratoria 78.000 anni fa.
Secondo Kivisild et al. (1999), "Le sovrapposizioni minori con i lignaggi descritti in altre popolazioni eurasiatiche dimostrano chiaramente che le recenti immigrazioni hanno avuto un impatto minimo sulla struttura innata del pool genetico materno degli asiatici del sud. Nonostante le variazioni riscontrate all'interno dell'India, queste popolazioni derivano da un limitato numero di lignaggi fondatori. Questi lignaggi furono probabilmente introdotti nell'Asia meridionale durante il Paleolitico medio, prima del popolamento dell'Europa 48.000 anni fa e forse del Vecchio Mondo in generale. Basu et al. (2003) sottolinea anche l'unità di fondo dei lignaggi femminili in India.
Y Variazione cromosomica
Le conclusioni basate sulla variazione del cromosoma Y sono state più varie di quelle basate sulla variazione del mtDNA. Mentre Kivisild et al. (2003) propone un patrimonio genetico antico e condiviso di lignaggi maschili nell'Asia meridionale, Bamshad et al. (2001) suggerisce un'affinità tra i lignaggi maschili dell'Asia meridionale e i moderni eurasiatici occidentali proporzionata al rango delle caste superiori e colloca le popolazioni delle caste superiori degli stati dell'India meridionale più vicine agli europei dell'est.
Basu et al. (2003) conclude che le popolazioni tribali austro-asiatiche sono entrate in India prima dal corridoio nord-occidentale e molto più tardi alcune di esse attraverso il corridoio nord-orientale. Considerando che Kumar et al. (2007) hanno analizzato 25 tribù austro-asiatiche dell'Asia meridionale e hanno trovato un forte legame genetico paterno tra i gruppi sublinguistici delle popolazioni austro-asiatiche dell'Asia meridionale. Mukherjee et al. (2001) colloca i pakistani e gli indiani del nord tra le popolazioni dell'Asia occidentale e dell'Asia centrale, mentre Cordaux et al. (2004) sostiene che le popolazioni di casta indiane sono più vicine alle popolazioni dell'Asia centrale. Saho et al. (2006) e Sengupta et al. (2006) suggeriscono che le popolazioni di casta indiane non sono state soggette a recenti mescolanze. Sanghamitra Sahoo conclude il suo studio con:
Non è necessario, sulla base delle prove attuali, guardare oltre l'Asia meridionale per le origini dell'eredità paterna della maggior parte degli indiani al momento dell'inizio dell'agricoltura stanziale. Il concetto perenne di persone, lingua e agricoltura che arrivano insieme in India attraverso il corridoio nord-occidentale non regge a un attento esame. Le recenti affermazioni per un collegamento degli aplogruppi J2, L, R1a e R2 con un'origine contemporanea per la maggior parte dei lignaggi paterni delle caste indiane al di fuori dell'Asia meridionale vengono respinte, sebbene i nostri risultati supportino un'origine locale degli aplogruppi F* e H. Degli altri, solo J2 indica un contributo esterno recente e inequivocabile, dall'Asia occidentale piuttosto che dall'Asia centrale. Le attuali distribuzioni delle frequenze degli aplogruppi sono, con l'eccezione dei lignaggi, prevalentemente guidate da determinanti geografiche, piuttosto che culturali. Ironia della sorte, è nel nord-est dell'India, tra i gruppi di tubercolosi, che ci sono prove evidenti di una diffusione demica su larga scala rintracciabile da geni, cultura e lingua, ma apparentemente non dall'agricoltura.
Analisi del vicino più vicino fatta da Mondal et al. nel 2017 ha concluso che i lignaggi Y indiani sono vicini alle popolazioni dell'Europa meridionale e al momento della divergenza tra le due migrazioni precedenti alla steppa:
Questi risultati suggeriscono che l'ascendenza legata all'Europa nelle popolazioni indiane potrebbe essere molto più antica e più complessa del previsto, e potrebbe provenire dalla prima ondata di agricoltori o anche prima.
— Mondal et al. 2017
Variazione autosomica del DNA
AASI-ANI-ASI
Anche i risultati degli studi basati sulla variazione del DNA autosomico sono stati variati. In un importante studio (2009) che utilizzava oltre 500.000 marcatori autosomici biallelici, Reich ipotizzò che la moderna popolazione dell'Asia meridionale fosse il risultato della commistione tra due popolazioni ancestrali geneticamente divergenti risalenti all'era post-Olocene. Questi due antichi popoli "ricostruiti" li chiamò "Ancestral South Indians" (ASI) e "Ancestral North Indians" (ANI). Secondo Reich: "L'ascendenza ANI è significativamente più alta negli indoeuropei rispetto ai parlanti dravidici, suggerendo che l'ancestrale ASI potrebbe aver parlato una lingua dravidica prima di mescolarsi con l'ANI". Mentre l'ANI è geneticamente vicino a mediorientali, centroasiatici ed europei, l'ASI non è strettamente correlato a gruppi al di fuori del subcontinente. Poiché non è disponibile alcun DNA antico "ASI", gli indigeni Andamanesi Onge sono usati come proxy (imperfetto) dell'ASI (secondo Reich et al., gli Andamanesi, sebbene distinti da loro, sono la popolazione vivente più vicina all'ASI). Secondo Reich et al., sia l'ascendenza ANI che ASI si trovano in tutto il subcontinente (sia nell'India settentrionale che meridionale) in proporzioni variabili e che "l'ascendenza ANI varia dal 39-71% in India, ed è più alta nell'area tradizionalmente superiore casta e parlanti indoeuropei."
Moorjani et al. Il 2013 afferma che l'ASI, sebbene non strettamente imparentato con nessun gruppo vivente, è "imparentato (lontano) con gli indigeni delle isole Andamane". Moorjani et al. tuttavia suggeriscono un possibile flusso genico nell'Andamanese da una popolazione imparentata con l'ASI, causando la relazione modellata. Lo studio ha concluso che "quasi tutti i gruppi che parlano lingue indoeuropee o dravidiche si trovano lungo un gradiente di varia parentela con gli eurasiatici occidentali nella PCA (denominato "clino indiano")".
Uno studio del 2013 di Chaubey che utilizza il polimorfismo a singolo nucleotide (SNP), mostra che il genoma del popolo andamanese (Onge) è più vicino a quello di altri gruppi di Negrito oceanici che a quello dei sud-asiatici.
Secondo Basu et al. 2016, ulteriori analisi hanno rivelato che la struttura genomica delle popolazioni indiane continentali è meglio spiegata dai contributi di quattro componenti ancestrali. Oltre all'ANI e all'ASI, Basu et. al (2016) ha identificato due componenti ancestrali dell'Asia orientale nell'India continentale che sono importanti per i tribali di lingua austro-asiatica e gli oratori tibeto-birmani, che hanno indicato come AAA (per "Ancestral Austro-Asiatic") e ATB (per " Ancestrale tibeto-birmano") rispettivamente. Lo studio deduce anche che le popolazioni dell'arcipelago delle Isole Andamane formano un'ascendenza distinta, che "è risultata essere coancestrali alle popolazioni oceaniche " ma più distanti dai sud-asiatici.
Il cline di commistione tra i ceppi ANI e ASI è datato al periodo del c. 4.2–1.9 kya di Moorjani et al. (2013), corrispondente all'età del bronzo indiana, e associato dagli autori al processo di deurbanizzazione della civiltà della valle dell'Indo e allo spostamento della popolazione al sistema Gangetico nell'incipiente età del ferro indiana. Basu et al. (2003) suggerisce che "i parlanti dravidici erano probabilmente diffusi in tutta l'India prima dell'arrivo dei nomadi di lingua indoeuropea" e che "la formazione di popolazioni per fissione che ha provocato effetti di fondazione e deriva hanno lasciato le loro impronte sulle strutture genetiche dei contemporanei popolazioni”. Il genetista PP Majumder (2010) ha recentemente sostenuto che i risultati di Reich et al. (2009) sono in notevole concordanza con la ricerca precedente utilizzando mtDNA e Y-DNA:
Si suppone che le popolazioni dell'Asia centrale abbiano contribuito maggiormente al pool genetico indiano, in particolare al pool genetico dell'India settentrionale, e che i migranti si fossero presumibilmente trasferiti in India attraverso quelli che oggi sono l'Afghanistan e il Pakistan. Utilizzando i dati sulla variazione del DNA mitocondriale raccolti da vari studi, abbiamo dimostrato che le popolazioni dell'Asia centrale e del Pakistan mostrano il coefficiente di differenziazione genetica più basso con le popolazioni dell'India settentrionale, una differenziazione più elevata con le popolazioni dell'India meridionale e la più alta con le popolazioni dell'India nord-orientale . Le popolazioni dell'India settentrionale sono geneticamente più vicine a quelle dell'Asia centrale rispetto alle popolazioni di altre regioni geografiche dell'India... . Coerentemente con i risultati di cui sopra, un recente studio che utilizza oltre 500.000 marcatori autosomici biallelici ha trovato un gradiente da nord a sud della vicinanza genetica delle popolazioni indiane agli eurasiatici occidentali. Questa caratteristica è probabilmente correlata alle proporzioni di antenati derivati dal pool genetico eurasiatico occidentale, che, come ha dimostrato questo studio, è maggiore nelle popolazioni che abitano l'India settentrionale rispetto a quelle che abitano l'India meridionale.
Chaubey et al. Il 2015 ha rilevato una caratteristica componente ancestrale dell'Asia orientale, principalmente limitata a popolazioni specifiche ai piedi dell'Himalaya e della parte nord-orientale dell'India. La maggiore frequenza della componente si osserva tra i gruppi di lingua tibeto-birmana dell'India nord-orientale ed è stata rilevata anche nelle popolazioni andamanesi al 32%, con presenza consistente anche tra i parlanti austroasiatici. Si trova in gran parte assente nei parlanti indoeuropei e dravidici, ad eccezione di alcuni gruppi etnici specifici che vivono ai piedi dell'Himalaya e dell'India centro-meridionale. I ricercatori hanno tuttavia suggerito che l'ascendenza dell'Asia orientale (rappresentata dagli Han) misurata nei gruppi andamanesi studiati potrebbe effettivamente riflettere la cattura dell'affinità degli andamanesi con i melanesiani e i negrito malesi (piuttosto che una vera mescolanza dell'Asia orientale), come uno studio precedente di Chaubey et al. suggeriva "una profonda discendenza comune" tra andamanesi, melanesiani e altri gruppi di negrito, e un'affinità tra negrito e melanesiani del sud-est asiatico (così come gli andamanesi) con gli asiatici orientali.
Lazaridis et al. (2016) osserva "L'impatto demografico delle popolazioni legate alle steppe sull'Asia meridionale è stato sostanziale, poiché i Mala , una popolazione Dalit dell'India meridionale con un minimo di antenati ancestrali dell'India settentrionale (ANI) hanno ancora ~ 18% di antenati Yamnaya legati alle steppe , mentre i Kalash del Pakistan si presume che abbia ~ 50% di discendenza Yamnaya correlata alle steppe, una delle percentuali più alte accanto agli europei dell'est al 51%." Lo studio del 2016 di Lazaridis et al. ha stimato (6,5-50,2%) la mescolanza correlata alle steppe negli asiatici del sud. Lazaridis et al. osserva inoltre che "Una direzione utile della ricerca futura è un campionamento più completo del DNA antico delle popolazioni della steppa, nonché delle popolazioni dell'Asia centrale (a est dell'Iran e a sud della steppa), che potrebbe rivelare fonti più vicine dell'ANI rispetto a quelli qui considerati, e dell'Asia meridionale per determinare direttamente la traiettoria del cambiamento della popolazione nell'area.
Pathak et al. Il 2018 ha concluso che gli oratori indoeuropei delle pianure del Gange e gli oratori dravidici hanno un'ascendenza significativa dell'età del bronzo medio-antica (Steppe_EMBA) di Yamnaya, ma nessuna discendenza della steppa dell'età del bronzo medio-tarda (Steppe_MLBA). D'altra parte, le popolazioni "indiane e pakistane nord-occidentali" (PNWI) hanno mostrato un'ascendenza significativa della Steppe_MLBA insieme a quella di Yamnaya (Steppe_EMBA). Lo studio ha anche notato che gli antichi campioni dell'Asia meridionale avevano Steppe_MLBA significativamente più alto di Steppe_EMBA (o Yamnaya). Lo studio ha anche suggerito che il Rors potrebbe essere utilizzato come proxy per l'ANI.
David Reich nel suo libro del 2018 Who We Are and How We Got Here afferma che le analisi del 2016 hanno rilevato che l'ASI ha quantità significative di una componente di ascendenza proveniente da agricoltori iraniani (circa il 25% dei loro antenati), con il restante 75% dei loro ascendenza derivante da cacciatori-raccoglitori nativi dell'Asia meridionale. Aggiunge che ASI era improbabile che i cacciatori-raccoglitori locali dell'Asia meridionale come precedentemente stabilito, ma una popolazione responsabile della diffusione dell'agricoltura in tutta l'Asia meridionale. Nel caso dell'ANI, l'ascendenza contadina iraniana è del 50%, mentre il resto proviene da gruppi della steppa legati allo Yamnaya.
Narasimhan et al. (2018) , allo stesso modo, concludono che ANI e ASI si sono formati nel II millennio a.C. Erano preceduti da un misto di AASI (antichi antenati dell'India meridionale, ovvero cacciatori-raccoglitori che condividevano una radice lontana con gli Andamanesi, gli Aborigeni australiani e gli asiatici orientali); e gli agricoltori iraniani che arrivarono in India ca. 4700-3000 aC, e "deve aver raggiunto la Valle dell'Indo entro il 4 ° millennio aC". Secondo Narasimhan et al., questa popolazione mista, che probabilmente era originaria della civiltà della valle dell'Indo, "contribuì in grandi proporzioni sia all'ANI che all'ASI", che prese forma durante il II millennio a.C. L'ANI è formato da un misto di " gruppi legati alla periferia dell'Indo " e migranti dalla steppa, mentre l'ASI è stato formato da " gruppi legati alla periferia dell'Indo " che si sono spostati a sud e si sono mescolati ulteriormente con i cacciatori-raccoglitori locali. Si suggerisce che l'ascendenza della popolazione ASI sia stata in media di circa il 73% dall'AASI e il 27% da agricoltori legati all'Iran. Narasimhan et al. osservare che i campioni del gruppo periferico dell'Indo sono sempre miscele delle stesse due fonti prossimali di AASI e antenati legati all'agricoltura iraniana; con "uno degli individui della periferia dell'Indo con ~ 42% di antenati AASI e gli altri due individui con ~ 14-18% di antenati AASI" (con il resto dei loro antenati provenienti dalla popolazione iraniana legata all'agricoltura). Gli autori propongono che i cacciatori-raccoglitori indigeni dell'AASI rappresentino un ramo divergente che si è separato all'incirca nello stesso periodo in cui gli antenati dell'Asia orientale, degli Onge (andamanesi) e degli aborigeni australiani si sono separati l'uno dall'altro. Ha dedotto, "essenzialmente tutta la discendenza degli odierni asiatici orientali e meridionali (prima della mescolanza correlata all'Eurasia occidentale negli asiatici meridionali) deriva da una singola diffusione verso est, che ha dato origine in un breve lasso di tempo ai lignaggi che portano all'AASI , asiatici orientali, Onge e australiani."
Uno studio genetico di Yelmen et al. (2019) tuttavia mostra che le moderne popolazioni dell'Asia meridionale sono generalmente più vicine tra loro e abbastanza distinte dalle popolazioni al di fuori dell'Asia meridionale. Hanno concluso che i moderni sud-asiatici sono fondamentalmente una miscela di una componente indigena dell'Asia meridionale (AASI) e di una componente eurasiatica occidentale (derivata sia dall'Asia occidentale che dalle steppe occidentali). Gli autori hanno anche rivelato che la componente indigena dell'Asia meridionale (AASI) è piuttosto distinta dagli andamanesi o degli asiatici orientali e che gli andamanesi sono quindi un proxy imperfetto. Propongono che il popolo tribale dell'India meridionale Paniya (un gruppo di ascendenza prevalentemente ASI) serva come sostituto migliore degli Andamanesi (Onge) per la componente "indigena dell'Asia meridionale" nei moderni sud asiatici, poiché i Paniya derivano direttamente dal nativi dell'Asia meridionale, mentre gli Onge potrebbero aver ricevuto il flusso genetico da altri gruppi. Hanno scoperto che la popolazione indigena dell'Asia meridionale era isolata da altre popolazioni almeno dal 45.000 aC.
Due studi genetici (Shinde et al. 2019 e Narasimhan et al. 2019) che analizzano i resti della civiltà della valle dell'Indo (di parti dell'India nordoccidentale e del Pakistan orientale dell'età del bronzo), hanno scoperto che avevano un misto di antenati, entrambi originari del sud Cacciatori-raccoglitori asiatici che condividono una radice lontana con gli Andamanesi e da un gruppo imparentato con agricoltori iraniani. I campioni analizzati da Shinde hanno derivato circa il 50-98% del loro genoma da popolazioni imparentate con l'Iran e dal 2-50% da cacciatori-raccoglitori nativi dell'Asia meridionale. I campioni analizzati da Narasimhan et al. aveva il 45-82% di antenati iraniani legati agli agricoltori e l'11-50% di origine di cacciatori-raccoglitori dell'Asia meridionale. I campioni analizzati di entrambi gli studi hanno poco o nessuno della componente " ascendenza delle steppe " associata alle successive migrazioni indoeuropee in India. Gli autori hanno scoperto che le rispettive quantità di tali antenati variavano in modo significativo tra gli individui e hanno concluso che sono necessari più campioni per ottenere il quadro completo della storia della popolazione indiana.
Distanza genetica tra gruppi di caste e tribù
Gli studi di Watkins et al. (2005) e Kivisild et al. (2003) sulla base di marcatori autosomici concludono che le caste indiane e le popolazioni tribali hanno un antenato comune. Reddy et al. (2005) hanno trovato distribuzioni di frequenza alleliche abbastanza uniformi tra i gruppi di caste dell'Andhra Pradesh meridionale , ma una distanza genetica significativamente maggiore tra gruppi di caste e tribù che indica l'isolamento genetico delle tribù e delle caste.
Viswanathan et al. (2004) in uno studio sulla struttura genetica e le affinità tra le popolazioni tribali dell'India meridionale conclude, " La differenziazione genetica era elevata e le distanze genetiche non erano significativamente correlate con le distanze geografiche. La deriva genetica quindi probabilmente ha giocato un ruolo significativo nel plasmare i modelli di variazione genetica osservata nelle popolazioni tribali dell'India meridionale.Altrimenti , le analisi dei rapporti di popolazione hanno mostrato che tutte le popolazioni indiane e dell'Asia meridionale sono ancora simili tra loro, indipendentemente dalle caratteristiche fenotipiche, e non mostrano particolari affinità con gli africani.Concludiamo che le somiglianze fenotipiche di alcuni gruppi indiani agli africani non riflettono una stretta relazione tra questi gruppi, ma sono meglio spiegati dalla convergenza ".
Uno studio del 2011 pubblicato sull'American Journal of Human Genetics indica che i componenti ancestrali indiani sono il risultato di una storia demografica più complessa di quanto si pensasse in precedenza. Secondo i ricercatori, l'Asia meridionale ospita due importanti componenti ancestrali, una delle quali è diffusa con frequenza e diversità genetica comparabili nelle popolazioni dell'Asia centrale, dell'Asia occidentale e dell'Europa; l'altra componente è più ristretta all'Asia meridionale. Tuttavia, se si dovesse escludere la possibilità di una migrazione indo-ariana su larga scala, questi risultati suggeriscono che le affinità genetiche di entrambi i componenti ancestrali indiani sono il risultato di molteplici flussi genici nel corso di migliaia di anni.
La modellizzazione delle diversità degli aplotipi osservate suggerisce che entrambi i componenti di origine indiana sono più antichi della presunta invasione indo-ariana di 3500 YBP. Coerentemente con i risultati delle distanze genetiche a coppie tra le regioni del mondo, gli indiani condividono più segnali di ascendenza con gli occidentali che con gli eurasiatici orientali.
Narashimhan et al 2019 hanno scoperto che i tribali Munda di lingua austroasiatica non potevano essere modellati semplicemente come una miscela di antenati ASI, AASI, ANI a differenza di altri sud-asiatici, ma richiedevano una componente di discendenza aggiuntiva dal sud-est asiatico. Sono stati modellati come una miscela dell'88% AASI e del 12% dell'Asia orientale legati al Nicobarese , quindi il profilo di ascendenza dei Munda fornisce una linea di discendenza indipendente dal sud-est asiatico intorno al 3° millennio a.C. Lipson et al. Il 2018 ha trovato risultati di mescolanza simili per quanto riguarda i tribali Munda affermando che "abbiamo ottenuto una buona corrispondenza con tre componenti di ascendenza: un eurasiatico occidentale, un eurasiatico orientale profondo (interpretato come un lignaggio indigeno dell'Asia meridionale) e uno del clade austroasiatico" . Lipson et al. Il 2018 ha inoltre scoperto che la fonte austroasiatica rivestita (proporzione del 35%) nei tribali Munda è stata dedotta come la più vicina a Mlabri . Allo stesso modo, Singh et al 2020 hanno scoperto che i parlanti austroasiatici nell'Asia meridionale escono dal cline dell'Asia meridionale a causa della loro affinità genetica del sud-est asiatico.
Guarda anche
- Archeogenetica
- Gruppi etnici dell'Asia meridionale
- Elenco delle regioni etnolinguistiche dell'Asia meridionale
- Popolazione dell'India
- Aplogruppi Y-DNA nelle popolazioni dell'Asia meridionale
- Studi genetici sui Gujarati
- Storia genetica dell'Europa
- Storia genetica del Medio Oriente
Appunti
Riferimenti
Ulteriori letture
- Allikas A, Ord D, Kurg R, Kivi S, Ustav M (giugno 2001). "Ruoli della regione cerniera e il dominio di legame del DNA della proteina E2 di tipo 1 del papillomavirus bovino nell'inizio della replicazione del DNA". Ricerca sui virus . 75 (2): 95-106. doi : 10.1016/S0168-1702(01)00219-2 . PMID 11325464 .
- Behar DM, Garrigan D, Kaplan ME, Mobasher Z, Rosengarten D, Karafet TM, et al. (marzo 2004). "Modelli contrastanti di variazione del cromosoma Y nelle popolazioni europee ebraiche ashkenazite e non ebree". Genetica Umana . 114 (4): 354–65. doi : 10.1007/s00439-003-1073-7 . PMID 14740294 . S2CID 10310338 .
- Bhattacharyya NP, Basu P, Das M, Pramanik S, Banerjee R, Roy B, et al. (agosto 1999). "Trascurabile flusso genico maschile attraverso i confini etnici in India, rivelato dall'analisi dei polimorfismi del DNA del cromosoma Y" . Ricerca sul genoma . 9 (8): 711-9. doi : 10.1101/gr.9.8.711 (inattivo 6 maggio 2021). PMID 10447506 .Manutenzione CS1: DOI inattivo da maggio 2021 ( link )
- Cann RL (marzo 2001). "Indizi genetici alla dispersione nelle popolazioni umane: ripercorrere il passato dal presente". Scienza . 291 (5509): 1742-8. Bibcode : 2001Sci...291.1742C . doi : 10.1126/science.1058948 . PMID 11249820 . S2CID 19367408 .
- Cinnioğlu C, King R, Kivisild T, Kalfoğlu E, Atasoy S, Cavalleri GL, et al. (gennaio 2004). "Scavo di strati aplotipici del cromosoma Y in Anatolia". Genetica Umana . 114 (2): 127-48. doi : 10.1007/s00439-003-1031-4 . PMID 14586639 . S2CID 10763736 .
- Das B, Chauhan PS, Seshadri M (ottobre 2004). "Condivisione minima di aplotipi STR del cromosoma Y tra cinque gruppi di popolazione endogama dell'India occidentale e sudoccidentale". Biologia Umana . 76 (5): 743-63. doi : 10.1353/hub.2005.0003 . PMID 15757245 . S2CID 12835244 .
- Hemphill BE, Christensen AF (3 novembre 1994). La civiltà dell'Oxus come collegamento tra Oriente e Occidente: un'analisi non metrica delle affinità biologiche del Bactrain dell'età del bronzo . Madison, Wisconsin. P. 13. (articolo letto alla South Asia Conference)
- Jobling MA, Tyler-Smith C (agosto 2003). "Il cromosoma Y umano: un marcatore evolutivo diventa maggiorenne". Recensioni sulla natura. Genetica . 4 (8): 598-612. doi : 10.1038/nrg1124 . PMID 12897772 . S2CID 13508130 .
- Kivisild T , Rootsi S, Metspalu M, Mastana S, Kaldma K, Parik J, et al. (febbraio 2003). "L'eredità genetica dei primi coloni persiste sia nelle popolazioni tribali indiane che in quelle di casta" . Giornale americano di genetica umana . 72 (2): 313-32. doi : 10.1086/346068 . PMC 379.225 . PMID 12536373 .
- Metspalu M, Kivisild T, Metspalu E, Parik J, Hudjashov G, Kaldma K, et al. (agosto 2004). "La maggior parte dei confini del mtDNA esistenti nell'Asia meridionale e sud-occidentale sono stati probabilmente modellati durante l'insediamento iniziale dell'Eurasia da umani anatomicamente moderni" . Genetica BMC . 5 : 26. doi : 10.1186/1471-2156-5-26 . PMC 516768 . PMID 15339343 .
- Patowary A, Purkanti R, Singh M, Chauhan RK, Bhartiya D, Dwivedi OP, et al. (luglio 2012). "Analisi sistematica e annotazione funzionale delle variazioni nel genoma di un individuo indiano". Mutazione umana . 33 (7): 1133–40. doi : 10.1002/humu.22091 . PMID 22461382 . S2CID 11466942 .
- Rootsi S, Magri C, Kivisild T, Benuzzi G, Help H, Bermisheva M, et al. (luglio 2004). "La filogeografia dell'aplogruppo I del cromosoma Y rivela domini distinti del flusso genico preistorico in Europa" . Giornale americano di genetica umana . 75 (1): 128-37. doi : 10.1086/422196 . PMC 1181996 . PMID 15162323 .
- Qamar R, Ayub Q, Mohyuddin A, Helgason A, Mazhar K, Mansoor A, et al. (maggio 2002). "Variazione del DNA cromosomico Y in Pakistan" . Giornale americano di genetica umana . 70 (5): 1107–24. doi : 10.1086/339929 . PMC 447.589 . PMID 11898125 .
- Semino O, Magri C, Benuzzi G, Lin AA, Al-Zahery N, Battaglia V, et al. (maggio 2004). "Origine, diffusione e differenziazione degli aplogruppi E e J del cromosoma Y: inferenze sulla neolitizzazione dell'Europa e successivi eventi migratori nell'area del Mediterraneo" . Giornale americano di genetica umana . 74 (5): 1023–34. doi : 10.1086/386295 . PMC 1181965 . PMID 15069642 .
Fonti
- Indian Genome Variation Consortium (aprile 2008). "Paesaggio genetico del popolo indiano: una tela per l'esplorazione del gene della malattia". Giornale di genetica . 87 (1): 3-20. doi : 10.1007/s12041-008-0002-x . PMID 18560169 . S2CID 21473349 .
- Endicott P, Metspalu M, Kivisild T (2007). "Prove genetiche sulle moderne dispersioni umane nell'Asia meridionale: cromosio Y e prospettive del DNA mitocondriale" . In Michael D. Petraglia, Bridget Allchin (a cura di). L'evoluzione e la storia delle popolazioni umane nell'Asia meridionale . Springer. pp. 201-228. ISBN 978-1-4020-5561-4.
- Hemphill B, Lukacs J, Kennedy K (1991). "Adattamenti biologici e affinità degli Harappans dell'età del bronzo". In Prato RH (ed.). Scavi di Harappa 1986-1990: un approccio multidisciplinare all'urbanistica del terzo millennio . pp. 137-82. ISBN 978-0-9629110-1-9.
- Kennedy KA (1984). "Una rivalutazione delle teorie delle origini razziali del popolo della civiltà della valle dell'Indo da dati antropologici recenti". In Kennedy KA, Possehl GL (a cura di). Studi di archeologia e paleoantropologia dell'Asia meridionale . Atlantic Highlands, NJ : Stampa umanistica. pp. 99-107.
- Kennedy KA (1995). "Sono stati identificati ariani nella documentazione scheletrica preistorica dell'Asia meridionale?" . In George Erdosy (ed.). Gli indoariani dell'antica Asia meridionale . Walter de Gruyter. pp. 49-54. ISBN 978-3-11-014447-5.
- Kivisild T (2000b). Le origini delle popolazioni eurasiatiche meridionali e occidentali: uno studio sul mtDNA (PDF) . Università di Tartu, Estonia. (dottorato)
- Mascarenhas DD, Raina A, Aston CE, Sanghera DK (2015). "Ricostruzione genetica e culturale della migrazione di un antico lignaggio" . BioMed Research International . 2015 : 651415. doi : 10.1155/2015/651415 . PMC 4605215 . PMID 26491681 .
- Narasimhan VM, Anthony D, Mallory J, Reich D (2018). La formazione genomica dell'Asia meridionale e centrale . bioRxiv 10.1101/292581 . doi : 10.1101/292581 . hdl : 21.11116/0000-0001-E7B3-0 . S2CID 89658279 .
- Oppenheimer S (2003). La vera vigilia: il viaggio dell'uomo moderno fuori dall'Africa . New York: Carroll e Graf Publishers. ISBN 978-0-7867-1192-5.
- Pamjav (dicembre 2012). "Breve comunicazione: i nuovi marcatori binari del cromosoma Y migliorano la risoluzione filogenetica all'interno dell'aplogruppo R1a1". Giornale americano di antropologia fisica . 149 (4): 611–615. doi : 10.1002/ajpa.22167 . PMID 23115110 . S2CID 4820868 .
- Renfrew C, Boyle K, ed. (2000a). Un'ascendenza indiana: una chiave per comprendere la diversità umana in Europa e oltre (PDF) . ISBN 978-1-902937-08-3.
- Silva M, Oliveira M, Vieira D, Brandão A, Rito T, Pereira JB, et al. (marzo 2017). "Una cronologia genetica per il subcontinente indiano indica dispersioni fortemente influenzate dal sesso" . Biologia Evolutiva BMC . 17 (1): 88. doi : 10.1186/s12862-017-0936-9 . PMC 5364613 . PMID 28335724 .
- Underhill PA (2003). "Inferire la storia umana: indizi dagli aplotipi del cromosoma Y". Simposi di Cold Spring Harbor sulla biologia quantitativa . 68 : 487–93. doi : 10.1101/sqb.2003.68.487 . PMID 15338652 . S2CID 15527457 .
- Underhill PA, Poznik GD, Rootsi S, Järve M, Lin AA, Wang J, et al. (gennaio 2015). "La struttura filogenetica e geografica dell'aplogruppo R1a del cromosoma Y" . Giornale europeo di genetica umana . 23 (1): 124-31. doi : 10.1038/ejhg.2014.50 . PMC 4266736 . PMID 24667786 .
- Wells S (2003). Il viaggio dell'uomo: un'odissea genetica . Princeton University Press. ISBN 9780691115320.
link esterno
- Introduzione agli aplogruppi e agli aplotipi , Mark A. Jobling, Università di Leicester. [1]
- Journey of Man: People of the World , Fondazione Bradshaw, in associazione con Stephen Oppenheimer .
- Indian Genome Variation Database Institute of Genomics and Integrative Biology
- Elenco delle frequenze R2
- Maldive




