Oncogenomica - Oncogenomics
L'oncogenomica è un sottocampo della genomica che caratterizza i geni associati al cancro . Si concentra sulle alterazioni genomiche, epigenomiche e della trascrizione nel cancro.
Il cancro è una malattia genetica causata dall'accumulo di mutazioni del DNA e alterazioni epigenetiche che portano alla proliferazione cellulare sfrenata e alla formazione di neoplasie . L'obiettivo dell'oncogenomica è identificare nuovi oncogeni o geni oncosoppressori che possano fornire nuove informazioni sulla diagnosi del cancro, prevedere l'esito clinico dei tumori e nuovi bersagli per le terapie antitumorali. Il successo delle terapie antitumorali mirate come Gleevec , Herceptin e Avastin ha sollevato la speranza per l'oncogenomica di chiarire nuovi bersagli per il trattamento del cancro.
Oltre a comprendere i meccanismi genetici sottostanti che avviano o guidano la progressione del cancro, l'oncogenomica si rivolge al trattamento del cancro personalizzato. Il cancro si sviluppa a causa di mutazioni del DNA e alterazioni epigenetiche che si accumulano casualmente. L'identificazione e il targeting delle mutazioni in un singolo paziente può portare a una maggiore efficacia del trattamento.
Il completamento del Progetto Genoma Umano ha facilitato il campo dell'oncogenomica e ha aumentato le capacità dei ricercatori di trovare gli oncogeni. Le tecnologie di sequenziamento e le tecniche di profilo di metilazione globale sono state applicate allo studio dell'oncogenomica.
Storia
L'era della genomica è iniziata negli anni '90, con la generazione di sequenze di DNA di molti organismi. Nel 21° secolo, il completamento del Progetto Genoma Umano ha consentito lo studio della genomica funzionale e l'esame dei genomi tumorali. Il cancro è un obiettivo principale.
L'era dell'epigenomica è iniziata in gran parte più di recente, intorno al 2000. Una delle principali fonti di cambiamento epigenetico è la metilazione alterata delle isole CpG nella regione del promotore dei geni (vedi metilazione del DNA nel cancro ). Un certo numero di metodi recentemente ideati può valutare lo stato di metilazione del DNA nei tumori rispetto ai tessuti normali. Alcuni metodi valutano la metilazione dei CpG situati in diverse classi di loci, comprese le isole, le coste e gli scaffali CpG, nonché i promotori, i corpi genici e le regioni intergeniche. Il cancro è anche uno dei principali obiettivi degli studi epigenetici.
L'accesso al sequenziamento dell'intero genoma del cancro è importante per la ricerca sul cancro (o sul genoma del cancro) perché:
- Le mutazioni sono la causa immediata del cancro e definiscono il fenotipo del tumore .
- L'accesso a campioni di tessuto canceroso e normale dello stesso paziente e il fatto che la maggior parte delle mutazioni cancerose rappresentino eventi somatici , consentono l'identificazione di mutazioni cancro-specifiche.
- Le mutazioni del cancro sono cumulative e talvolta sono correlate allo stadio della malattia. Si distinguono metastasi e resistenza ai farmaci.
L'accesso al profilo di metilazione è importante per la ricerca sul cancro perché:
- Gli epi-driver, insieme ai Mut-driver, possono agire come cause immediate di cancro
- Le epimutazioni del cancro sono cumulative e talvolta correlate allo stadio della malattia
Sequenziamento dell'intero genoma
Il primo genoma del cancro è stato sequenziato nel 2008. Questo studio ha sequenziato un tipico genoma di leucemia mieloide acuta (AML) e il suo genoma normale controparte ottenuto dallo stesso paziente. Il confronto ha rivelato dieci geni mutati. Si pensava già che due contribuissero alla progressione del tumore: una duplicazione interna in tandem del gene della tirosina chinasi del recettore FLT3 , che attiva la segnalazione della chinasi ed è associata a una prognosi sfavorevole e un inserimento di quattro basi nell'esone 12 del gene NPM1 (NPMc). Queste mutazioni si trovano nel 25-30% dei tumori AML e si pensa che contribuiscano alla progressione della malattia piuttosto che causarla direttamente.
Le restanti 8 erano nuove mutazioni e tutte erano cambiamenti di base singola: quattro erano in famiglie fortemente associate alla patogenesi del cancro ( PTPRT , CDH24, PCLKC e SLC15A1 ). Gli altri quattro non avevano alcuna precedente associazione con la patogenesi del cancro. Avevano potenziali funzioni nelle vie metaboliche che suggerivano meccanismi attraverso i quali potevano agire per promuovere il cancro (KNDC1, GPR124 , EB12, GRINC1B)
Questi geni sono coinvolti in percorsi noti per contribuire alla patogenesi del cancro, ma prima di questo studio la maggior parte non sarebbe stata candidata per la terapia genica mirata. Questa analisi ha convalidato l'approccio del sequenziamento dell'intero genoma del cancro nell'identificazione delle mutazioni somatiche e l'importanza del sequenziamento parallelo dei genomi delle cellule normali e tumorali.
Nel 2011 è stato sequenziato il genoma di un eccezionale malato di cancro alla vescica il cui tumore era stato eliminato dal farmaco everolimus , rivelando mutazioni in due geni, TSC1 e NF2 . Le mutazioni hanno disregolato mTOR , la proteina inibita da everolimus, permettendole di riprodursi senza limiti. Di conseguenza, nel 2015 è stata creata l'iniziativa Exceptional Responders presso il National Cancer Institute. L'iniziativa consente a questi pazienti eccezionali (che hanno risposto positivamente per almeno sei mesi a un farmaco antitumorale che di solito fallisce) di far sequenziare i loro genomi per identificare le mutazioni rilevanti. Una volta identificati, altri pazienti potrebbero essere sottoposti a screening per tali mutazioni e quindi ricevere il farmaco. Nel 2016 A tal fine, nel 2015 è iniziata una sperimentazione nazionale sui farmaci antitumorali che ha coinvolto fino a ventiquattrocento centri. I pazienti con mutazioni appropriate sono abbinati a uno degli oltre quaranta farmaci.
Nel 2014 il Center for Molecular Oncology ha lanciato il test MSK-IMPACT, uno strumento di screening che cerca mutazioni in 341 geni associati al cancro. Nel 2015 erano stati sottoposti a screening più di cinquemila pazienti. I pazienti con mutazioni appropriate possono essere arruolati in studi clinici che forniscono una terapia mirata.
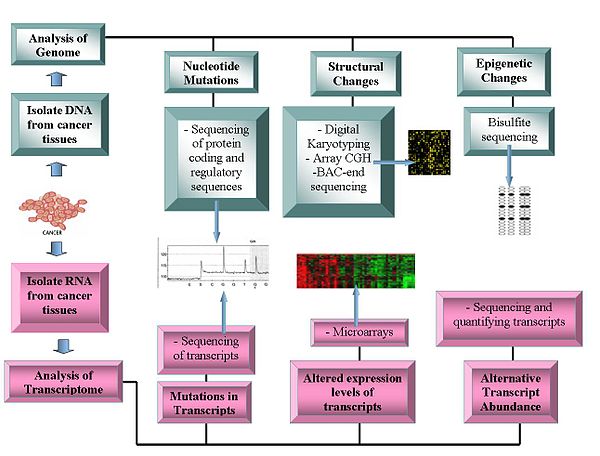
Tecnologie
Le tecnologie genomiche includono:
Sequenziamento del genoma
- Sequenziamento del DNA : Pyrosequencing -based sequencer offrono un metodo relativamente basso costo per generare dati di sequenza.
- Ibridazione comparativa del genoma dell'array : questa tecnica misura ledifferenze del numero di copie del DNA tra genomi normali e cancerosi. Utilizza l'intensità di fluorescenza di campioni marcati con fluorescenza, che vengono ibridati a sonde note su un microarray.
- Analisi rappresentazionale del microarray di oligonucleotidi : rileva la variazione del numero di copie utilizzando frammenti genomici amplificati digeriti per restrizione che vengono ibridati a oligonucleotidi umani, raggiungendo una risoluzione compresa tra 30 e 35 kbit/s.
- Cariotipizzazione digitale : rileva la variazione del numero di copie utilizzando tag genomici ottenuti tramite digest con enzimi di restrizione . Questi tag vengono quindi collegati a ditag, concatenati, clonati, sequenziati e mappati di nuovo al genoma di riferimento per valutare la densità dei tag.
- Sequenziamento dell'estremità del cromosoma artificiale batterico (BAC) ( profilo della sequenza finale ) : identifica ibreakpoint cromosomici generando una libreria BAC da un genoma del cancro e sequenziando le loro estremità. I cloni BAC che contengono aberrazioni cromosomiche hanno sequenze finali che non si mappano a una regione simile del genoma di riferimento, identificando così un punto di rottura cromosomico.
trascrittomi
- Microarray : valuta l' abbondanza dei trascritti . Utile nella classificazione, nella prognosi, aumenta la possibilità di approcci terapeutici differenziali e aiuta l'identificazione di mutazioni nelle regioni codificanti delle proteine. La relativa abbondanza di trascrizioni alternative è diventata una caratteristica importante della ricerca sul cancro. Particolari forme di trascrizione alternative sono correlate a specifici tipi di cancro.
- RNA-Seq
Bioinformatica e analisi funzionale degli oncogeni
Le tecnologie bioinformatiche consentono l'analisi statistica dei dati genomici. Le caratteristiche funzionali degli oncogeni devono ancora essere stabilite. Le potenziali funzioni includono le loro capacità di trasformazione relative alla formazione del tumore e ruoli specifici in ogni fase dello sviluppo del cancro.
Dopo il rilevamento di mutazioni del cancro somatico in una coorte di campioni di cancro, è possibile eseguire analisi computazionali bioinformatiche per identificare probabili mutazioni funzionali e probabili driver. Esistono tre approcci principali abitualmente utilizzati per questa identificazione: mappare le mutazioni, valutare l'effetto della mutazione della funzione di una proteina o di un elemento regolatorio e trovare segni di selezione positiva in una coorte di tumori. Gli approcci non sono necessariamente sequenziali, tuttavia, esistono importanti relazioni di precedenza tra elementi dei diversi approcci. Diversi strumenti vengono utilizzati in ogni fase.
operemia
L'operomica mira a integrare genomica, trascrittomica e proteomica per comprendere i meccanismi molecolari che stanno alla base dello sviluppo del cancro.
Oncogenomica comparata
L'oncogenomica comparativa utilizza confronti tra specie per identificare gli oncogeni. Questa ricerca prevede lo studio di genomi, trascrittomi e proteomi del cancro in organismi modello come i topi, l'identificazione di potenziali oncogeni e il riferimento a campioni di cancro umano per vedere se gli omologhi di questi oncogeni sono importanti nel causare tumori umani. Le alterazioni genetiche nei modelli murini sono simili a quelle riscontrate nei tumori umani. Questi modelli sono generati da metodi che includono la mutagenesi per inserzione retrovirale o il trapianto di innesto di cellule cancerose.
Fonte di mutazioni cancerogene, mutagenesi del cancro
Le mutazioni forniscono la materia prima per la selezione naturale in evoluzione e possono essere causate da errori di replicazione del DNA, dall'azione di mutageni esogeni o da danni endogeni al DNA. Il meccanismo di replicazione e mantenimento del genoma può essere danneggiato da mutazioni o alterato da condizioni fisiologiche e livelli differenziali di espressione nel cancro (vedi riferimenti in ).
Come sottolineato da Gao et al., la stabilità e l'integrità del genoma umano sono mantenute dal sistema di risposta al danno del DNA (DDR). Il danno non riparato al DNA è una delle principali cause di mutazioni che guidano la carcinogenesi. Se la riparazione del DNA è carente, il danno al DNA tende ad accumularsi. Tale danno al DNA in eccesso può aumentare gli errori mutazionali durante la replicazione del DNA a causa della sintesi di traslezione soggetta a errori . Il danno eccessivo al DNA può anche aumentare le alterazioni epigenetiche dovute a errori durante la riparazione del DNA. Tali mutazioni e alterazioni epigenetiche possono dare origine al cancro . I geni DDR sono spesso repressi nel cancro umano da meccanismi epigenetici. Tale repressione può comportare la metilazione del DNA delle regioni del promotore o la repressione dei geni DDR da parte di un microRNA. La repressione epigenetica dei geni DDR si verifica più frequentemente della mutazione genica in molti tipi di cancro (vedi Epigenetica del cancro ). Pertanto, la repressione epigenetica gioca spesso un ruolo più importante della mutazione nel ridurre l'espressione dei geni DDR. Questa ridotta espressione dei geni DDR è probabilmente un importante fattore di carcinogenesi.
Il contesto della sequenza nucleotidica influenza la probabilità di mutazione e l'analisi dei motivi del DNA mutazionale (mutabile) può essere essenziale per comprendere i meccanismi della mutagenesi nel cancro. Tali motivi rappresentano le impronte digitali delle interazioni tra DNA e mutageni, tra DNA ed enzimi di riparazione/replicazione/modifica. Esempi di motivi sono il motivo AID WRCY/RGYW (W = A o T, R = purina e Y = pirimidina) con mutazioni da C a T/G/A, e mutazioni soggette a errore DNA pol η attribuite a AID (da A a G/C/G) nei motivi WA/TW.
Un altro modo (agnostico) per analizzare gli spettri mutazionali osservati e il contesto della sequenza del DNA delle mutazioni nei tumori prevede il raggruppamento di tutte le mutazioni di diversi tipi e contesti dai campioni di cancro in una distribuzione discreta. Se sono disponibili più campioni di cancro, le loro mutazioni dipendenti dal contesto possono essere rappresentate sotto forma di una matrice non negativa. Questa matrice può essere ulteriormente scomposta in componenti (firme mutazionali) che idealmente dovrebbero descrivere i singoli fattori mutageni. Diversi metodi di calcolo sono stati proposti per risolvere questo problema di decomposizione. La prima implementazione del metodo NMF (Non-negative Matrix Factorization) è disponibile nel Sanger Institute Mutational Signature Framework sotto forma di un pacchetto MATLAB. D'altra parte, se sono disponibili solo mutazioni da un singolo campione di tumore, il pacchetto DeconstructSigs R e il server MutaGene possono fornire l'identificazione dei contributi di diverse firme mutazionali per un singolo campione di tumore. Inoltre, il server MutaGene fornisce modelli mutageni o mutazionali specifici per il cancro e firme che possono essere applicati per calcolare la mutabilità prevista del DNA e del sito proteico per disaccoppiare i contributi relativi di mutagenesi e selezione nella carcinogenesi.
letalità sintetica
La letalità sintetica si verifica quando una combinazione di carenze nell'espressione di due o più geni porta alla morte cellulare, mentre una carenza in uno solo di questi geni non lo fa. Le carenze possono derivare da mutazioni, alterazioni epigenetiche o inibitori di uno dei geni.
Il potenziale terapeutico della letalità sintetica come efficace strategia antitumorale è in continuo miglioramento. Recentemente, l'applicabilità della letalità sintetica alla terapia antitumorale mirata è aumentata grazie al recente lavoro di scienziati tra cui Ronald A. DePinho e colleghi, in quella che viene definita "letalità collaterale". Muller et al. hanno scoperto che i geni passeggeri, con la vicinanza cromosomica ai geni oncosoppressori, sono eliminati in modo collaterale in alcuni tumori. Pertanto, l'identificazione di geni ridondanti deleti collaterali che svolgono una funzione cellulare essenziale può essere il serbatoio non sfruttato per perseguire un approccio di letalità sintetica . La letalità collaterale ha quindi un grande potenziale nell'identificazione di nuovi bersagli terapeutici selettivi in oncologia. Nel 2012, Muller et al. identificato che la delezione omozigote del gene ENO1 glicolitico essenziale ridondante nel glioblastoma umano (GBM) è la conseguenza della vicinanza alle delezioni del locus soppressore del tumore 1p36 e può contenere il potenziale per un approccio di letalità sintetica all'inibizione del GBM. ENO1 è uno dei tre geni omologhi ( ENO2 , ENO3 ) che codifica per l' enzima alfa-enolasi dei mammiferi . ENO2 , che codifica per l' enolasi 2 , è principalmente espresso nei tessuti neurali, portando alla postulazione che nel GBM deleto con ENO1 , ENO2 possa essere il bersaglio ideale come omologo ridondante di ENO1 . Muller ha scoperto che l' inibizione di ENO2 sia genetica che farmacologica nelle cellule GBM con delezione omozigote di ENO1 provoca un risultato di letalità sintetica mediante l'uccisione selettiva delle cellule GBM. Nel 2016, Muller e colleghi hanno scoperto SF2312 antibiotico come nanomolari-gamma estremamente potente enolasi inibitore che la proliferazione di cellule di glioma preferenzialmente inibisce e flusso glicolitico in ENO1 -deleted cellule. SF2312 ha dimostrato di essere più efficace dell'inibitore della pan-enolasi PhAH e ha una maggiore specificità per l' inibizione di ENO2 rispetto a ENO1 . Il lavoro successivo dello stesso team ha mostrato che lo stesso approccio potrebbe essere applicato al cancro del pancreas , per cui la delezione omozigote SMAD4 porta alla delezione collaterale dell'enzima malico mitocondriale 2 ( ME2 ), una decarbossilasi ossidativa essenziale per l' omeostasi redox . Dey et al. mostrano che la delezione genomica di ME2 nelle cellule di adenocarcinoma duttale pancreatico si traduce in specie reattive dell'ossigeno endogene, coerenti con il cancro del pancreas guidato da KRAS e essenzialmente innesca le cellule ME2-null per la letalità sintetica mediante l'esaurimento dell'isoforma ME3 dipendente da NAD(P)+ ridondante. È stato scoperto che gli effetti della deplezione di ME3 sono mediati dall'inibizione della sintesi de novo nucleotidi risultante dall'attivazione di AMPK e dall'apoptosi mitocondriale mediata da ROS. Nel frattempo, Oike et al. hanno dimostrato la generalizzabilità del concetto prendendo di mira geni essenziali ridondanti in processi diversi dal metabolismo, vale a dire le subunità SMARCA4 e SMARCA2 nel complesso SWI/SNF di rimodellamento della cromatina .
Alcuni oncogeni sono essenziali per la sopravvivenza di tutte le cellule (non solo delle cellule cancerose). Pertanto, i farmaci che eliminano questi oncogeni (e quindi uccidono le cellule tumorali) possono anche danneggiare le cellule normali, inducendo malattie significative. Tuttavia, altri geni possono essere essenziali per le cellule cancerose ma non per le cellule sane.
I trattamenti basati sul principio della letalità sintetica hanno prolungato la sopravvivenza dei malati di cancro e sono promettenti per futuri progressi nell'inversione della carcinogenesi. Un tipo importante di letalità sintetica opera sul difetto di riparazione del DNA che spesso avvia un cancro ed è ancora presente nelle cellule tumorali. Alcuni esempi sono forniti qui.
L' espressione di BRCA1 o BRCA2 è carente nella maggior parte dei tumori di alto grado della mammella e dell'ovaio, solitamente a causa della metilazione epigenetica del suo promotore o della repressione epigenetica da parte di un microRNA sovraespresso (vedi articoli BRCA1 e BRCA2 ). BRCA1 e BRCA2 sono componenti importanti della via principale per la riparazione ricombinante omologa delle rotture del doppio filamento. Se uno o l'altro è carente, aumenta il rischio di cancro, in particolare il cancro al seno o alle ovaie. Un percorso di riparazione del DNA di backup, per alcuni dei danni solitamente riparati da BRCA1 e BRCA2, dipende da PARP1 . Pertanto, molti tumori ovarici rispondono a un trattamento approvato dalla FDA con un inibitore di PARP, causando letalità sintetica per le cellule tumorali carenti di BRCA1 o BRCA2. Questo trattamento è in fase di valutazione anche per il cancro al seno e numerosi altri tumori in studi clinici di fase III nel 2016.
Esistono due vie per la riparazione ricombinante omologa delle rotture del doppio filamento. La via principale dipende da BRCA1 , PALB2 e BRCA2, mentre una via alternativa dipende da RAD52. Studi preclinici, che coinvolgono cellule epigeneticamente ridotte o mutate di BRCA (in coltura o iniettate nei topi), mostrano che l'inibizione di RAD52 è sinteticamente letale con la carenza di BRCA.
Le mutazioni nei geni impiegati nella riparazione del disadattamento del DNA (MMR) causano un alto tasso di mutazione. Nei tumori, tali frequenti mutazioni successive spesso generano antigeni immunogeni "non self". Uno studio clinico di fase II sull'uomo, con 41 pazienti, ha valutato un approccio letale sintetico per i tumori con o senza difetti MMR. Il prodotto del gene PD-1 normalmente reprime le risposte immunitarie citotossiche. L'inibizione di questo gene consente una maggiore risposta immunitaria. Quando i pazienti oncologici con un difetto di MMR nei loro tumori sono stati esposti a un inibitore di PD-1, il 67% - 78% dei pazienti ha avuto una sopravvivenza libera da progressione correlata al sistema immunitario. Al contrario, per i pazienti senza MMR difettoso, l'aggiunta dell'inibitore PD-1 ha generato solo l'11% dei pazienti con sopravvivenza libera da progressione correlata al sistema immunitario. Quindi l'inibizione di PD-1 è principalmente sinteticamente letale con difetti MMR.
ARID1A , un modificatore della cromatina, è necessario per l' unione delle estremità non omologhe , una via principale che ripara le rotture del doppio filamento nel DNA e ha anche ruoli regolatori della trascrizione. Le mutazioni ARID1A sono una delle 12 mutazioni cancerogene più comuni. La mutazione o l'espressione epigeneticamente ridotta di ARID1A è stata trovata in 17 tipi di cancro. Studi preclinici nelle cellule e nei topi mostrano che la letalità sintetica per il deficit di ARID1A si verifica mediante l'inibizione dell'attività della metiltransferasi di EZH2, o con l'aggiunta dell'inibitore della chinasi dasatinib.
Un altro approccio consiste nell'eliminare individualmente ciascun gene in un genoma e osservare l'effetto sulle cellule normali e cancerose. Se il knockout di un gene altrimenti non essenziale ha poco o nessun effetto sulle cellule sane, ma è letale per le cellule cancerose contenenti un oncogene mutato, allora la soppressione a livello di sistema del gene soppresso può distruggere le cellule cancerose lasciando quelle sane relativamente intatte. La tecnica è stata utilizzata per identificare gli inibitori di PARP-1 per il trattamento dei tumori associati a BRCA1 / BRCA2 . In questo caso, la presenza combinata dell'inibizione di PARP-1 e delle mutazioni associate al cancro nei geni BRCA è letale solo per le cellule cancerose.
Banche dati per la ricerca sul cancro
Il Cancer Genome Project è un'iniziativa per mappare tutte le mutazioni somatiche nel cancro. Il progetto sequenzia sistematicamente gli esoni e le giunzioni di giunzione fiancheggianti dei genomi dei tumori primari e delle linee cellulari cancerose. Il software COSMIC visualizza i dati generati da questi esperimenti. A febbraio 2008, il CGP aveva identificato 4.746 geni e 2.985 mutazioni in 1.848 tumori.
Il progetto Cancer Genome Anatomy include informazioni sulla ricerca sui genomi, trascrittomi e proteomi del cancro.
Progenetix è un database di riferimento oncogenomico, che presenta dati sui tumori citogenetici e citogenetici molecolari.
Oncomine ha raccolto dati dai profili del trascrittoma del cancro.
Il database oncogenomico integrativo IntOGen e i set di dati Gitools integrano dati oncogenomici umani multidimensionali classificati per tipo di tumore. La prima versione di IntOGen si è concentrata sul ruolo dell'espressione genica deregolata e della CNV nel cancro. Una versione successiva ha enfatizzato i geni responsabili del cancro mutazionale in 28 tipi di tumore. Tutte le versioni dei dati IntOGen sono rese disponibili nel database IntOGen.
L' International Cancer Genome Consortium è il più grande progetto per raccogliere dati sul genoma del cancro umano. I dati sono accessibili attraverso il sito web dell'ICGC. La BioExpress® Oncology Suite contiene dati di espressione genica da campioni di tumori primari, metastatici e benigni e campioni normali, compresi i controlli adiacenti abbinati. La suite include campioni di tumori maligni ematologici per molti tumori noti.
Database specifici per animali modello includono il Retrovirus Tagged Cancer Gene Database (RTCGD) che ha raccolto ricerche sulla mutagenesi inserzionale retrovirale e trasposonica nei tumori del topo.
Famiglie geniche
L'analisi mutazionale di intere famiglie di geni ha rivelato che i geni della stessa famiglia hanno funzioni simili, come previsto da sequenze codificanti e domini proteici simili . Due di queste classi sono la famiglia della chinasi , coinvolta nell'aggiunta di gruppi fosfato alle proteine e la famiglia della fosfatasi , coinvolta nella rimozione dei gruppi fosfato dalle proteine. Queste famiglie sono state esaminate per la prima volta a causa del loro ruolo apparente nella trasduzione dei segnali cellulari di crescita o morte cellulare. In particolare, più del 50% dei tumori del colon-retto porta una mutazione in un gene della chinasi o della fosfatasi. Il gene della fosfatidilinositold 3-chinasi ( PIK3CA ) codifica per le chinasi lipidiche che comunemente contengono mutazioni a carico del colon-retto, della mammella, dello stomaco, del polmone e di vari altri tumori. Le terapie farmacologiche possono inibire PIK3CA. Un altro esempio è il gene BRAF , uno dei primi ad essere implicato nei melanomi. BRAF codifica per una serina / treonina chinasi coinvolta nella via di segnalazione della crescita RAS-RAF- MAPK . Le mutazioni in BRAF causano fosforilazione costitutiva e attività nel 59% dei melanomi. Prima del BRAF, il meccanismo genetico dello sviluppo del melanoma era sconosciuto e quindi la prognosi per i pazienti era sfavorevole.
DNA mitocondriale
Le mutazioni del DNA mitocondriale (mtDNA) sono legate alla formazione di tumori. Sono stati identificati quattro tipi di mutazioni del mtDNA:
mutazioni puntiformi
Sono state osservate mutazioni puntiformi nella regione codificante e non codificante del mtDNA contenuto nelle cellule tumorali. Negli individui con tumori della vescica, della testa/collo e del polmone, le mutazioni puntiformi all'interno della regione codificante mostrano segni di somiglianza. Ciò suggerisce che quando una cellula sana si trasforma in una cellula tumorale (una trasformazione neoplastica) i mitocondri sembrano diventare omogenei. Mutazioni puntiformi abbondanti situate all'interno della regione non codificante, D-loop , dei mitocondri cancerosi suggeriscono che le mutazioni all'interno di questa regione potrebbero essere una caratteristica importante in alcuni tumori.
Eliminazioni
Questo tipo di mutazione viene rilevata sporadicamente a causa delle sue piccole dimensioni (< 1kb). La comparsa di alcune mutazioni specifiche del mtDNA (delezione di 264 bp e delezione di 66 bp nel gene complesso 1 subunità ND1) in più tipi di cancro fornisce alcune prove che piccole delezioni del mtDNA potrebbero apparire all'inizio della tumorigenesi . Suggerisce anche che la quantità di mitocondri contenenti queste delezioni aumenta man mano che il tumore progredisce. Un'eccezione è una delezione relativamente grande che appare in molti tumori (nota come "delezione comune"), ma sono state trovate più delezioni su larga scala del mtDNA nelle cellule normali rispetto alle cellule tumorali. Ciò può essere dovuto a un processo apparentemente adattativo delle cellule tumorali per eliminare qualsiasi mitocondrio che contiene queste delezioni su larga scala (la "delezione comune" è > 4kb).
Inserzioni
Due piccole inserzioni di mtDNA di ~260 e ~520 bp possono essere presenti nel cancro al seno, nel cancro gastrico, nel carcinoma epatocellulare (HCC) e nel cancro del colon e nelle cellule normali. Non è stata stabilita alcuna correlazione tra questi inserimenti e il cancro.
Copia mutazioni del numero
La caratterizzazione del mtDNA tramite saggi di reazione a catena della polimerasi in tempo reale mostra la presenza di alterazioni quantitative del numero di copie del mtDNA in molti tumori. Si prevede che si verifichi un aumento del numero di copie a causa dello stress ossidativo. D'altra parte, si pensa che la diminuzione sia causata da mutazioni puntiformi somatiche nel sito di origine della replicazione del filamento H e/o del c-stretch omopolimerico D310 nella regione dell'ansa D, mutazioni nel gene p53 (gene soppressore del tumore) via mediata e/o attività enzimatica inefficiente a causa di mutazioni POLG . Qualsiasi aumento/diminuzione del numero di copie rimane quindi costante all'interno delle cellule tumorali. Il fatto che la quantità di mtDNA sia costante nelle cellule tumorali suggerisce che la quantità di mtDNA è controllata da un sistema molto più complicato nelle cellule tumorali, piuttosto che semplicemente alterata come conseguenza di una proliferazione cellulare anormale. Il ruolo del contenuto di mtDNA nei tumori umani apparentemente varia per particolari tipi o siti di tumore.
| Tipo di cancro | Posizione delle mutazioni puntiformi | Posizione nucleotidica delle delezioni | Aumento della copia mtDNA # | Diminuzione della copia mtDNA # | |||
|---|---|---|---|---|---|---|---|
| D-Loop | mRNA | tRNA | rRNA | ||||
| Vescia | X | X | X | 15,642-15.662 | |||
| Seno | X | X | X | X | 8470-13.447 e 8482-13459 | X | |
| Testa e collo | X | X | X | X | 8470-13.447 e 8482-13459 | X | |
| Orale | X | X | 8470-13.447 e 8482-13459 | ||||
| Carcinoma epatocellulare (HCC) | X | X | X | X | 306-556 e 3894-3960 | X | |
| Esofageo | X | X | X | 8470-13.447 e 8482-13459 | X | ||
| Gastrico | X | X | X | 298-348 | X | ||
| Prostata | X | X | 8470-13.447 e 8482-13459 | X | |||
Il 57,7% (500/867) conteneva punti somatici e delle 1172 mutazioni esaminate il 37,8% (443/1127) era localizzato nella regione di controllo del D-loop, il 13,1% (154/1172) era localizzato nei geni tRNA o rRNA e Il 49,1% (575/1127) è stato trovato nei geni mRNA necessari per la produzione di complessi necessari per la respirazione mitocondriale.
Applicazioni diagnostiche
Alcuni farmaci antitumorali prendono di mira il mtDNA e hanno mostrato risultati positivi nell'uccidere le cellule tumorali. La ricerca ha utilizzato le mutazioni mitocondriali come biomarcatori per la terapia delle cellule tumorali. È più facile indirizzare la mutazione all'interno del DNA mitocondriale rispetto al DNA nucleare perché il genoma mitocondriale è molto più piccolo e più facile da esaminare per mutazioni specifiche. Le alterazioni del contenuto di MtDNA riscontrate nei campioni di sangue potrebbero essere in grado di fungere da marker di screening per prevedere la futura suscettibilità al cancro e per monitorare la progressione del tumore maligno. Insieme a queste potenziali caratteristiche utili del mtDNA, non è sotto il controllo del ciclo cellulare ed è importante per mantenere la generazione di ATP e l'omeostasi mitocondriale. Queste caratteristiche rendono il targeting del mtDNA una pratica strategia terapeutica.
Biomarcatori del cancro
Diversi biomarcatori possono essere utili nella stadiazione, nella prognosi e nel trattamento del cancro. Possono variare da polimorfismi a singolo nucleotide (SNP), aberrazioni cromosomiche , cambiamenti nel numero di copie del DNA, instabilità dei microsatelliti, metilazione della regione del promotore o anche livelli proteici alti o bassi.
Guarda anche
- Oncogenomica personalizzata
- Oncotecture
- L'atlante del genoma del cancro
- Consorzio Internazionale Genoma Cancro