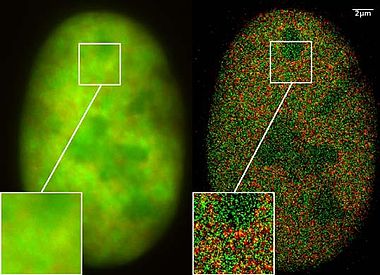
Illuminazione a modulazione spaziale Vertico - Vertico spatially modulated illumination
L'illuminazione a modulazione spaziale Vertico ( Vertico-SMI ) è il microscopio ottico più veloce per l'analisi 3D di cellule complete nell'intervallo nanometrico . Si basa su due tecnologie sviluppate nel 1996, SMI (illuminazione a modulazione spaziale) e SPDM (microscopia a distanza di precisione spettrale). La risoluzione ottica effettiva di questo nanoscopio ottico ha raggiunto la prossimità di 5 nm in 2D e 40 nm in 3D, superando notevolmente il limite di risoluzione λ / 2 (circa 200 nm per la luce blu) applicato alla microscopia standard che utilizza la trasmissione o la riflessione della luce naturale (al contrario dell'illuminazione strutturata o della fluorescenza ) secondo il limite di risoluzione di Abbe Questo limite (noto anche come limite di Rayleigh ) era stato determinato da Ernst Abbe nel 1873 e regola il limite di risoluzione raggiungibile dei microscopi utilizzando tecniche convenzionali.
Il microscopio Vertico-SMI è stato sviluppato da un team guidato da Christoph Cremer , emerito presso l'Università di Heidelberg , e si basa sulla combinazione di tecniche ottiche leggere di microscopia di localizzazione (SPDM, microscopia a distanza di precisione spettrale ) e illuminazione strutturata (SMI, illuminazione a modulazione spaziale ).
Da marzo 2008 molti coloranti fluorescenti standard come GFP e coloranti fluorescenti Alexa possono essere utilizzati con questa microscopia di localizzazione cosiddetta SPDMphymod (fluorofori modificabili fisicamente), per la quale solo una singola lunghezza d'onda laser di intensità adeguata è sufficiente per il nanoimaging.
Configurazione
SMI sta per un tipo speciale di illuminazione ottica laser (illuminazione a modulazione spaziale ) e Vertico riflette la disposizione verticale dell'asse del microscopio che rende possibile l'analisi di cellule fisse ma anche di cellule viventi con una risoluzione ottica inferiore a 10 nanometri (1 nanometro = 1 nm = 1 × 10 −9 m).
Una particolarità di questa tecnologia rispetto alle tecniche di focalizzazione come la microscopia 4Pi , sono le esposizioni ad ampio campo che consentono di rappresentare intere cellule su nanoscala. Una tale esposizione 3D di un'intera cellula con una dimensione tipica dell'oggetto di 20 µm × 20 µm richiede solo 2 minuti. Le esposizioni ad ampio campo indicano che l'intero oggetto viene illuminato e rilevato simultaneamente.
Illuminazione a modulazione spaziale
La microscopia SMI è un processo ottico leggero della cosiddetta funzione di diffusione del punto -ingegneria. Si tratta di processi che modificano la funzione di diffusione del punto (PSF) di un microscopio in modo appropriato per aumentare la risoluzione ottica, per massimizzare la precisione delle misurazioni della distanza di oggetti fluorescenti che sono piccoli rispetto alla lunghezza d' onda della luce illuminante, o per estrarre altri parametri strutturali nell'intervallo nanometrico.
Il microscopio SMI in fase di sviluppo presso il Kirchhoff Institute for Physics presso l'Università di Heidelberg raggiunge questo obiettivo nel modo seguente: L'intensità di illuminazione all'interno del campo dell'oggetto non è uniforme, a differenza dei microscopi a fluorescenza a campo ampio convenzionali, ma è modulata spazialmente in modo preciso dall'uso di due fasci laser interferenti opposti lungo l'asse. Il principio del campo d'onda modulato spazialmente è stato sviluppato nel 1993 da Bailey et al. L'approccio microscopico SMI utilizzato nell'applicazione di Heidelberg sposta l'oggetto in passi di alta precisione attraverso il campo d'onda, oppure il campo d'onda stesso viene spostato rispetto all'oggetto mediante spostamento di fase. Ciò si traduce in una dimensione assiale e una risoluzione della distanza migliorate.
SMI può essere combinato con altre tecnologie di super risoluzione, ad esempio con 3D LIMON o LSI- TIRF come interferometro a riflessione interna totale con illuminazione strutturata lateralmente. Questa tecnica SMI ha permesso di acquisire immagini ottiche di luce delle distribuzioni di autofluoroforo nelle sezioni del tessuto dell'occhio umano con una risoluzione ottica senza precedenti. L'utilizzo di tre diverse lunghezze d'onda di eccitazione (488, 568 e 647 nm), consente di raccogliere informazioni spettrali sul segnale di autofluorescenza. Questo è stato utilizzato per il tessuto dell'occhio umano affetto da degenerazione maculare AMD.
SPDM: microscopia di localizzazione
Una singola, minuscola sorgente di luce può essere localizzata molto meglio della risoluzione di un microscopio: sebbene la luce produca un punto sfocato, è possibile utilizzare algoritmi informatici per calcolare con precisione il centro del punto sfocato, tenendo conto della funzione di diffusione del punto del microscopio, le proprietà di rumore del rivelatore e così via. Tuttavia, questo approccio non funziona quando ci sono troppe sorgenti vicine l'una all'altra: le sorgenti si sfocano tutte insieme.
SPDM (microscopia a distanza di precisione spettrale) è una famiglia di tecniche di microscopia a fluorescenza che aggira questo problema misurando solo poche sorgenti alla volta, in modo che ciascuna sorgente sia "otticamente isolata" dalle altre (cioè, separata da più del risoluzione del microscopio, tipicamente ~ 200-250 nm). Quindi, è possibile utilizzare la tecnica sopra (trovare il centro di ogni punto sfocato).
Se le molecole hanno una varietà di spettri diversi (spettri di assorbimento e / o spettri di emissione), è possibile osservare la luce da poche molecole alla volta utilizzando le sorgenti luminose e i filtri appropriati. Le molecole possono anche essere distinte in modi più sottili in base alla durata della fluorescenza e ad altre tecniche.
La risoluzione strutturale ottenibile utilizzando SPDM può essere espressa in termini di minima distanza misurabile tra due nella loro posizione spaziale determinata particella puntiforme di differenti caratteristiche spettrali ("risoluzione topologica"). La modellizzazione ha dimostrato che in condizioni adeguate per quanto riguarda la precisione di localizzazione, densità di particelle ecc., La "risoluzione topologica" corrisponde ad una " frequenza spaziale " che in termini di definizione classica è equivalente a una risoluzione ottica molto migliorata.
SPDM è una microscopia di localizzazione che raggiunge una risoluzione ottica effettiva molte volte migliore rispetto alla risoluzione ottica convenzionale (circa 200-250 nm), rappresentata dalla metà della larghezza del massimo principale della funzione di immagine puntiforme effettiva. Applicando opportuni processi di precisione ottica laser, la posizione e le distanze significativamente inferiori alla metà della larghezza della funzione di diffusione del punto (convenzionalmente 200-250 nm) possono essere misurate con precisione nanometrica tra target con diverse firme spettrali. Un'importante area di applicazione è la ricerca sul genoma (studio dell'organizzazione funzionale del genoma ). Un'altra importante area di utilizzo è la ricerca sulla struttura delle membrane.
Una delle basi più importanti della microscopia di localizzazione in generale è il primo lavoro sperimentale per la localizzazione di oggetti fluorescenti in nanoscala (3D) nel 1996 e la prova teorica e sperimentale per una precisione di localizzazione utilizzando la luce visibile nell'intervallo di 1 nm - la base per la microscopia di localizzazione migliore di 1/100 della lunghezza d'onda.
SPDMphymod: coloranti fluorescenti standard in modalità lampeggiante come GFP
Solo negli ultimi due anni sono state utilizzate negli studi nanoscopici molecole che emettono la stessa frequenza di luce spettrale (ma con firme spettrali diverse in base alle caratteristiche del lampeggiamento) ma che possono essere accese e spente per mezzo della luce come necessario per la precisione spettrale microscopia a distanza. Combinando molte migliaia di immagini della stessa cella, è stato possibile utilizzare misurazioni di precisione ottica laser per registrare immagini di localizzazione con una risoluzione ottica notevolmente migliorata. L'applicazione di questi nuovi processi di nanoscopia è apparsa fino a poco tempo fa molto difficile perché si presumeva che solo le molecole prodotte appositamente potessero essere accese e spente in modo appropriato utilizzando la luce.
Nel marzo 2008 il laboratorio di Christoph Cremer ha scoperto che ciò era possibile anche per molti coloranti fluorescenti standard come GFP , coloranti Alexa e molecole di fluoresceina, a condizione che fossero presenti determinate condizioni foto-fisiche. Utilizzando questa cosiddetta tecnologia SPDMphymod (fluorofori modificabili fisicamente), una singola lunghezza d'onda del laser di intensità adeguata è sufficiente per il nanoimaging. Al contrario, altre microscopie di localizzazione richiedono due lunghezze d'onda laser quando vengono utilizzate speciali molecole di fluorescenza foto-commutabili / fotoattivabili.
Il gene GFP è stato introdotto ed espresso in molte cellule procariotiche ed eucariotiche e il Premio Nobel per la Chimica 2008 è stato assegnato a Martin Chalfie , Osamu Shimomura e Roger Y. Tsien per la loro scoperta e lo sviluppo della proteina fluorescente verde. La scoperta che queste molecole fluorescenti standard possono essere utilizzate estende l'applicabilità del metodo SPMD a numerosi campi di ricerca in biofisica , biologia cellulare e medicina .
Coloranti fluorescenti standard già utilizzati con successo con la tecnologia SPDMphymod: GFP, RFP, YFP, Alexa 488, Alexa 568, Alexa 647, Cy2, Cy3, Atto 488 e fluoresceina.
LIMON: microscopia 3D a super risoluzione
LIMON (Light MicrOscopical nanosizing microscopy) è stato inventato nel 2001 presso l'Università di Heidelberg e combina la microscopia di localizzazione e l'illuminazione a modulazione spaziale alla microscopia a super risoluzione 3D.
Le immagini 3D che utilizzano Vertico-SMI sono rese possibili dalla combinazione di SMI e SPDM, per cui viene applicato prima il processo SMI e poi il processo SPDM. Il processo SMI determina il centro delle particelle e la loro diffusione nella direzione dell'asse del microscopio. Mentre il centro delle particelle / molecole può essere determinato con una precisione di 1-2 nm, la diffusione attorno a questo punto può essere determinata fino a un diametro assiale di ca. 30-40 nm.
Successivamente, la posizione laterale delle singole particelle / molecole viene determinata utilizzando SPDM, ottenendo una precisione di pochi nanometri. Attualmente, SPDM raggiunge 16 frame / sec con una risoluzione effettiva di 10 nm in 2D (piano dell'oggetto); circa 2000 fotogrammi di questo tipo vengono combinati con i dati SMI (tempo di acquisizione di circa 10 secondi) per ottenere un'immagine tridimensionale di massima risoluzione (risoluzione ottica 3D effettiva di circa 40-50 nm). Con una fotocamera più veloce , ci si può aspettare velocità ancora più elevate (fino a diverse centinaia di fotogrammi / sec, in fase di sviluppo). Utilizzando coloranti adatti, dovrebbero essere possibili risoluzioni ottiche 3D effettive ancora più elevate
Combinando SPDMphymod con SMI (entrambi inventati nel laboratorio di Christoph Cremer nel 1996) è stata ottenuta una ricostruzione 3D a doppio colore delle disposizioni spaziali dei cluster Her2 / neu e Her3. Le posizioni in tutte e tre le direzioni dei cluster di proteine potrebbero essere determinate con una precisione di circa 25 nm.
Uso della microscopia a super risoluzione nell'industria
Nonostante il suo utilizzo nei laboratori biomedici, le tecnologie di super risoluzione potrebbero servire come strumenti importanti nella ricerca farmaceutica. Potrebbero essere particolarmente utili per l'identificazione e la valutazione degli obiettivi. Ad esempio, le macchine biomolecolari (BMM) sono nanostrutture altamente complesse costituite da diverse molecole di grandi dimensioni e che sono responsabili delle funzioni di base nelle cellule del corpo. A seconda del loro stato funzionale, hanno una struttura 3D definita. Esempi di macchine biomolecolari sono i nucleosomi che consentono al DNA, un portatore di informazioni genetiche lungo due metri, di piegarsi nelle cellule del corpo in uno spazio di solo pochi milionesimi di millimetro di diametro. Pertanto, il DNA può fungere da centro di informazione e controllo.
Utilizzando LIMON 3D in combinazione con l'etichettatura del complesso LIMON, è possibile per la prima volta rendere visibili le proteine nascoste o gli acidi nucleici di un complesso di molecole 3D delle cosiddette macchine biomolecolari senza distruggere il complesso. Fino ad ora, il problema nella maggior parte dei casi era che il complesso doveva essere distrutto per un'analisi dettagliata delle singole macromolecole al suo interno. In alternativa, sono stati utilizzati modelli di simulazione al computer virtuale o costosi metodi di risonanza magnetica nucleare per visualizzare la struttura tridimensionale di tali complessi.
Letteratura
link esterno
- Superrisoluzione GFP
- Elenco delle pubblicazioni nanoscopia ottica
- comunicato stampa dell'Università di Heidelberg