SDHB - SDHB
La succinato deidrogenasi [ubichinone] subunità ferro-zolfo, mitocondriale (SDHB) nota anche come subunità ferro-zolfo del complesso II (Ip) è una proteina che nell'uomo è codificata dal gene SDHB .
Il complesso proteico succinato deidrogenasi (chiamato anche SDH o Complex II) catalizza l'ossidazione del succinato (succinato + ubichinone => fumarato + ubiquinolo). SDHB è una delle quattro subunità proteiche che formano la succinato deidrogenasi, le altre tre sono SDHA , SDHC e SDHD . La subunità SDHB è collegata alla subunità SDHA sull'estremità idrofila catalitica del complesso SDH. È anche connesso alle subunità SDHC / SDHD sull'estremità idrofobica del complesso ancorato nella membrana mitocondriale. La subunità è una proteina ferro-zolfo con tre cluster ferro-zolfo. Pesa 30 kDa .
Struttura
Il gene che codifica per la proteina SDHB è il DNA nucleare , non mitocondriale . Tuttavia, la proteina espressa si trova nella membrana interna dei mitocondri . La posizione del gene nell'uomo è sul primo cromosoma nel locus p36.1-p35. Il gene è codificato in 1.162 paia di basi, partizionate in 8 esoni . La proteina espressa pesa 31,6 kDa ed è composta da 280 aminoacidi. SDHB contiene i cluster ferro-zolfo necessari per il tunneling degli elettroni attraverso il complesso. Si trova tra SDHA e le due subunità transmembrana SDHC e SDHD .
Funzione
Il complesso SDH si trova sulla membrana interna dei mitocondri e partecipa sia al ciclo dell'acido citrico che alla catena respiratoria . SDHB agisce come un intermedio nell'azione dell'enzima SDH di base mostrata nella Figura 1:
- L'SDHA converte il succinato in fumarato come parte del ciclo dell'acido citrico . Questa reazione converte anche FAD in FADH 2 .
- Gli elettroni dal FADH 2 vengono trasferiti ai cluster di ferro della subunità SDHB [2Fe-2S],[4Fe-4S],[3Fe-4S].
- Infine gli elettroni vengono trasferiti al pool di Ubichinone (Q) tramite le subunità SDHC / SDHD . Questa funzione fa parte della catena respiratoria .
Inizialmente, SDHA ossida il succinato tramite deprotonazione nel sito di legame del FAD, formando FADH 2 e lasciando il fumarato , liberamente legato al sito attivo, libero di uscire dalla proteina. Gli elettroni da FADH 2 vengono trasferiti ai cluster di ferro della subunità SDHB [2Fe-2S],[4Fe-4S],[3Fe-4S] e si incanalano lungo il relè [Fe-S] fino a raggiungere lo zolfo di ferro [3Fe-4S] grappolo . Gli elettroni vengono quindi trasferiti a una molecola di ubichinone in attesa presso il sito attivo del pool Q nel dimero SDHC / SDHD . L' ossigeno carbonilico O1 dell'ubichinone è orientato nel sito attivo (immagine 4) dalle interazioni del legame idrogeno con Tyr83 di SDHD . La presenza di elettroni nel cluster ferro-zolfo [3Fe-4S] induce il movimento dell'ubichinone in un secondo orientamento. Ciò facilita una seconda interazione legame idrogeno tra il gruppo carbonilico O4 di ubichinone e Ser27 di SDHC . Dopo la prima fase di riduzione di un singolo elettrone, si forma una specie di radicale semichinone . Il secondo elettrone arriva dal cluster [3Fe-4S] per fornire una riduzione completa dell'ubichinone a ubiquinolo .
Significato clinico
Mutazioni germinali nel gene possono causare paraganglioma familiare (nella vecchia nomenclatura, Paraganglioma tipo PGL4). La stessa condizione è spesso chiamata feocromocitoma familiare . Meno frequentemente, il carcinoma a cellule renali può essere causato da questa mutazione.
I paragangliomi correlati alle mutazioni SDHB hanno un alto tasso di malignità. Quando è maligno, il trattamento è attualmente lo stesso di qualsiasi paraganglioma/feocromocitoma maligno.
Cancro
I paragangliomi causati da mutazioni SDHB hanno diverse caratteristiche distintive:
- La malignità è comune e varia dal 38% all'83% nei portatori con malattia. Al contrario, i tumori causati da mutazioni SDHD sono quasi sempre benigni. I paragangliomi sporadici sono maligni in meno del 10% dei casi.
- I paragangliomi maligni causati da SDHB sono solitamente (forse il 92%) extra-surrenalici. I feocromocitomi/paragangliomi sporadici sono extra-surrenalici in meno del 10% dei casi.
- La penetranza del gene è spesso riportata come 77% all'età di 50 anni (cioè il 77% dei portatori avrà almeno un tumore all'età di 50 anni). Questa è probabilmente una sopravvalutazione. Attualmente (2011), le famiglie con mutazioni SDHB silenti vengono sottoposte a screening per determinare la frequenza dei portatori silenti.
- L'età media di insorgenza è approssimativamente la stessa per la malattia SDHB rispetto a quella non correlata a SDHB (approssimativamente 36 anni).
Sono state osservate mutazioni che causano la malattia negli esoni da 1 a 7, ma non 8. Come con i geni SDHC e SDHD , SDHB è un gene oncosoppressore .
La formazione del tumore segue generalmente l' ipotesi del "due colpi" di Knudson . La prima copia del gene è mutata in tutte le cellule, tuttavia la seconda copia funziona normalmente. Quando la seconda copia muta in una determinata cellula a causa di un evento casuale, si verifica la perdita di eterozigosi (LOH) e la proteina SDHB non viene più prodotta. La formazione del tumore diventa quindi possibile.
Data la natura fondamentale della proteina SDH in tutte le funzioni cellulari, attualmente non è chiaro perché solo le cellule paragangliari siano interessate. Tuttavia, la sensibilità di queste cellule ai livelli di ossigeno può svolgere un ruolo.
Percorsi della malattia
Il percorso preciso che porta dalla mutazione SDHB alla tumorigenesi non è determinato; ci sono diversi meccanismi proposti.
Generazione di specie reattive dell'ossigeno
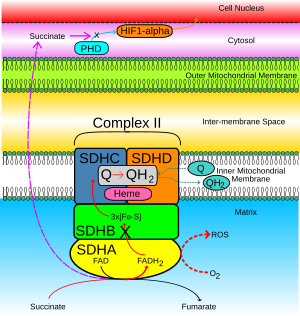
Quando l'attività del succinato-ubichinone viene inibita, gli elettroni che normalmente si trasferirebbero attraverso la subunità SDHB al pool di ubichinone vengono invece trasferiti all'O 2 per creare specie reattive dell'ossigeno (ROS) come il superossido . La freccia rossa tratteggiata nella Figura 2 lo mostra. I ROS accumulano e stabilizzano la produzione di HIF1-α . HIF1-α si combina con HIF1-β per formare il complesso eterodimerico HIF stabile, che a sua volta porta all'induzione di geni antiapoptotici nel nucleo cellulare.
Accumulo di succinato nel citosol
L'inattivazione dell'SDH può bloccare l'ossidazione del succinato , avviando una cascata di reazioni:
- Il succinato accumulato nella matrice mitocondriale si diffonde attraverso le membrane mitocondriali interna ed esterna al citosol (frecce tratteggiate viola nella Figura 2).
- In condizioni di normale funzione cellulare, HIF1-α nel citosol viene rapidamente idrossilato dalla prolil idrossilasi (PHD), mostrato con la freccia azzurra. Questo processo è bloccato dal succinato accumulato.
- HIF1-α si stabilizza e passa al nucleo cellulare (freccia arancione) dove si combina con HIF1-β per formare un complesso HIF attivo che induce l'espressione dei geni che causano il tumore.
Questo percorso aumenta la possibilità di un trattamento terapeutico. L'accumulo di succinato inibisce l'attività di PHD. L'azione della PHD richiede normalmente ossigeno e alfa-chetoglutarato come cosubstrati e ferro ferroso e ascorbato come cofattori . Il succinato compete con l'α-chetoglutarato nel legarsi all'enzima PHD. Pertanto, l'aumento dei livelli di α-chetoglutarato può compensare l'effetto dell'accumulo di succinato.
Il normale α-chetoglutarato non permea le pareti cellulari in modo efficiente ed è necessario creare un derivato permeante cellulare (es. esteri di α-chetoglutarato). Gli studi in vitro mostrano che questo approccio di integrazione può ridurre i livelli di HIF1-α e può portare a un approccio terapeutico ai tumori derivanti dalla carenza di SDH.
Apoptosi dello sviluppo alterata
Il tessuto paragangliare deriva dalle cellule della cresta neurale presenti in un embrione . Le cellule paragangliari extra-surrenaliche addominali secernono catecolamine che svolgono un ruolo importante nello sviluppo fetale. Dopo la nascita queste cellule di solito muoiono, un processo innescato da un declino del fattore di crescita nervoso (NGF) che avvia l' apoptosi (morte cellulare).
Questo processo di morte cellulare è mediato da un enzima chiamato prolil idrossilasi EglN3. L'accumulo di succinato causato dall'inattivazione di SDH inibisce la prolil idrossilasi EglN3. Il risultato netto è che il tessuto parangliare che normalmente morirebbe dopo la nascita rimane, e questo tessuto potrebbe essere in grado di innescare il paraganglioma/feocromocitoma in seguito.
Aumento della glicolisi
L'inibizione del ciclo dell'acido citrico costringe la cellula a creare ATP glicoliticamente per generare l'energia necessaria. Gli enzimi glicolitici indotti potrebbero potenzialmente bloccare l'apoptosi cellulare.
Modifica dell'RNA
Le trascrizioni dell'mRNA del gene SDHB nell'uomo sono modificate attraverso un meccanismo sconosciuto nella posizione del nucleotide ORF 136 causando la conversione di C in U e generando così un codone di stop con conseguente traduzione delle trascrizioni modificate in una proteina SDHB troncata con un amino R46X cambio acido. Questo editing è stato mostrato nei monociti e in alcune linee cellulari linfoidi umane ed è potenziato dall'ipossia .
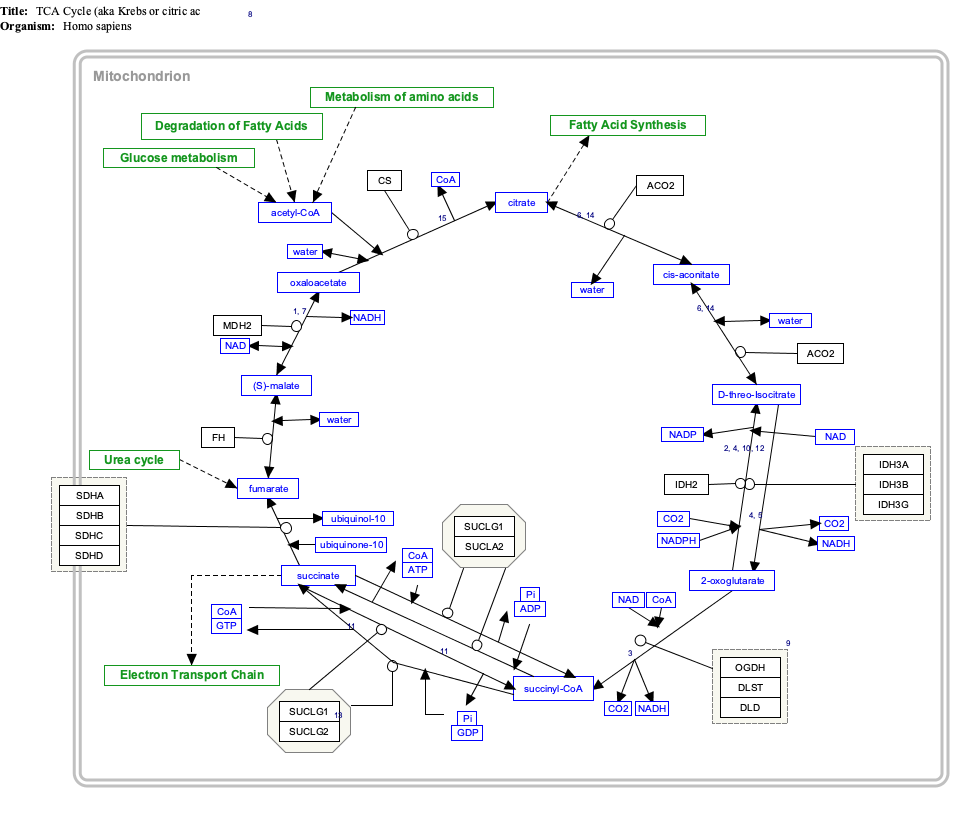
Mappa interattiva dei percorsi
Fare clic su geni, proteine e metaboliti di seguito per collegarsi ai rispettivi articoli.
Riferimenti
Ulteriori letture
- Milosevic D, Lundquist P, Cradic K, et al. (2010). "Sviluppo e convalida di un test completo di rilevamento di mutazioni e delezioni per SDHB, SDHC e SDHD" . Clin. Biochimica . 43 (7-8): 700-4. doi : 10.1016/j.clinbiochem.2010.01.016 . PMC 3419008 . PMID 20153743 .
- Alrashdi I, Bano G, Maher ER, Hodgson SV (2010). "Triade di Carney contro la sindrome di Carney Stratakis: due casi che illustrano la difficoltà nel distinguere tra queste condizioni nei singoli pazienti". Fam. Cancro . 9 (3): 443-7. doi : 10.1007/s10689-010-9323-z . PMID 20119652 . S2CID 21792188 .
- Okada Y, Kamatani Y, Takahashi A, et al. (2010). "Uno studio di associazione sull'intero genoma in 19 633 soggetti giapponesi ha identificato LHX3-QSOX2 e IGF1 come loci dell'altezza degli adulti" . Ronzio. Mol. Genetta . 19 (11): 2303-12. doi : 10.1093/hmg/ddq091 . PMID 20189936 .
- Bayley JP (2010). "Queste mutazioni eterozigoti composte di SDHB sono davvero mutazioni?". Cancro del sangue pediatrico . 55 (1): 211, risposta dell'autore 212. doi : 10.1002/pbc.22455 . PMID 20213850 . S2CID 31378042 .
- Rose JE, Behm FM, Drgon T, et al. (2010). "Cessazione personalizzata del fumo: interazioni tra dose di nicotina, dipendenza e punteggio del genotipo per smettere di fumare" . Mol. Med . 16 (7-8): 247-53. doi : 10.2119/molmed.2009.00159 . PMC 2896464 . PMID 20379614 .
- Gill AJ, Benn DE, Chou A, et al. (2010). "Immunoistochimica per SDHB triage test genetici di SDHB, SDHC e SDHD nelle sindromi paraganglioma-feocromocitoma". Ronzio. Patolo . 41 (6): 805–14. doi : 10.1016/j.humpath.2009.12.005 . PMID 20236688 .
- Martin TP, Irving RM, Maher ER (2007). "La genetica dei paragangliomi: una rassegna" . Clin Otorinolaringoiatria . 32 (1): 7-11. doi : 10.1111/j.1365-2273.2007.01378.x . PMID 17298303 .
- Eng C, Kiuru M, Fernandez MJ, Aaltonen LA (2003). "Un ruolo per gli enzimi mitocondriali nelle neoplasie ereditarie e oltre". Naz. Rev. Cancro . 3 (3): 193-202. doi : 10.1038/nrc1013 . PMID 12612654 . S2CID 20549458 .
- Lee J, Wang J, Torbenson M, et al. (2010). "Perdita di geni SDHB e NF1 in un tumore filloide maligno del seno come rilevato dall'ibridazione genomica comparativa oligo-array". Genet Cancro. Citogeneto . 196 (2): 179-83. doi : 10.1016/j.cancergencyto.2009.09.005 . PMID 20082856 .
- Hermsen MA, Siviglia MA, Llorente JL, et al. (2010). "Rilevanza dello screening delle mutazioni germinali nel paraganglioma della testa e del collo sia familiare che sporadico per la diagnosi precoce e la gestione clinica" . Cellula. Oncol . 32 (4): 275-83. doi : 10.3233/CLO-2009-0498 . PMC 4619289 . PMID 20208144 .
- Musil Z; Puchmajerová A; Krepelová A; et al. (2010). "Paraganglioma in una ragazza di 13 anni: una nuova mutazione del gene SDHB in famiglia?". Genet Cancro. Citogeneto . 197 (2): 189-92. doi : 10.1016/j.cancergencyto.2009.11.010 . PMID 20193854 .
- Shimada M, Miyagawa T, Kawashima M, et al. (2010). "Un approccio basato su uno studio di associazione genome-wide rivela loci candidati per la narcolessia". Ronzio. Genetta . 128 (4): 433–41. doi : 10.1007/s00439-010-0862-z . PMID 20677014 . S2CID 24207887 .
- Brière JJ; Favier J; El Ghouzzi V; et al. (2005). "Deficit di succinato deidrogenasi nell'uomo". Cellula. Mol. Vita Sci . 62 (19-20): 2317-24. doi : 10.1007/s00018-005-5237-6 . PMID 16143825 . S2CID 23793565 .
- Schimke RN, Collins DL, Stolle CA (2010). "Paraganglioma, neuroblastoma e una mutazione SDHB: risoluzione di un mistero di 30 anni". Sono. J. Med. Genet. un . 152A (6): 1531–5. doi : 10.1002/ajmg.a.33384 . PMID 20503330 . S2CID 22768946 .
- Gill AJ, Chou A, Vilain R, et al. (2010). "Immunoistochimica per SDHB divide i tumori stromali gastrointestinali (GIST) in 2 tipi distinti". Sono. J. Surg. Patolo . 34 (5): 636-44. doi : 10.1097/PAS.0b013e3181d6150d . PMID 20305538 . S2CID 2314622 .
-
Hendrickson SL, Lautenberger JA, Chinn LW, et al. (2010). "Varianti genetiche nei geni mitocondriali codificati dal nucleare influenzano la progressione dell'AIDS" . PLOS UNO . 5 (9): e12862. Bibcode : 2010PLoSO...512862H . doi : 10.1371/journal.pone.0012862 . PMC 2943476 . PMID 20877624 .

- Cerecer-Gil NY, Figuera LE, Llamas FJ, et al. (2010). "La mutazione di SDHB è una causa di paraganglioma d'alta quota correlato all'ipossia" . Clin. Ris . Cancro . 16 (16): 4148-54. doi : 10.1158/1078-0432.CCR-10-0637 . PMID 20592014 .
- Krawczyk A, Hasse-Lazar K, Pawlaczek A, et al. (2010). "Mutazioni germinali dei geni RET, SDHB, SDHD e VHL in pazienti con feocromocitomi e paragangliomi apparentemente sporadici". Endokrynol Pol . 61 (1): 43-8. PMID 20205103 .
-
Hes FJ, Weiss MM, Woortman SA, et al. (2010). "Bassa penetranza di una mutazione SDHB in una grande famiglia di paragangliomi olandesi" . BMC Med. Genetta . 11 : 92. doi : 10.1186/1471-2350-11-92 . PMC 2891715 . PMID 20540712 .

- Bailey SD, Xie C, Do R, et al. (2010). "La variazione al locus NFATC2 aumenta il rischio di edema indotto da tiazolidinedione nello studio Diabetes REduction Assessment con ramipril e rosiglitazone Medication (DREAM)" . Cura del diabete . 33 (10): 2250-3. doi : 10.2337/dc10-0452 . PMC 2945168 . PMID 20628086 .
-
Baysal BE (2007). "Una mutazione ricorrente del codone di arresto nel gene della subunità B della succinato deidrogenasi nel sangue periferico normale e nella leucemia acuta delle cellule T dell'infanzia" . PLOS UNO . 2 (5): e436. Bibcode : 2007PLoSO...2..436B . doi : 10.1371/journal.pone.0000436 . PMC 1855983 . PMID 17487275 .

- Baysal BE, Jong KD, Liu B, et al. (2013). "L'editing dell'RNA codificante C-to-U inducibile dall'ipossia downregola SDHB nei monociti" . PeerJ . 1 : e152. doi : 10.7717/peerj.152 . PMC 3775634 . PMID 24058882 .