Atromentin - Atromentin
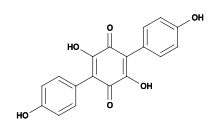 Formula strutturale di atromentin
|
|
| Nomi | |
|---|---|
|
Nome IUPAC preferito
1 4 , 2 3 , 2 6 , 3 4 -Tetraidrossi [1 1 , 2 1 : 2 4 , 3 1- trifenile] -2 2 , 2 5 -dione |
|
| Altri nomi
2,5-Diidrossi-3,6-bis (4-idrossifenil) -1,4-benzochinone
|
|
| Identificatori | |
|
Modello 3D ( JSmol )
|
|
| ChEBI | |
| ChemSpider | |
|
PubChem CID
|
|
| UNII | |
|
CompTox Dashboard ( EPA )
|
|
|
|
|
|
| Proprietà | |
| C 18 H 12 O 6 | |
| Massa molare | 324,288 g · mol −1 |
|
Salvo dove diversamente indicato, i dati sono forniti per i materiali nel loro stato standard (a 25 ° C [77 ° F], 100 kPa). |
|
|
|
|
| Riferimenti di Infobox | |
L'atromentin è un composto chimico naturale che si trova nei funghi Agaricomycetes negli ordini Agaricales e Thelephorales . Può anche essere preparato mediante sintesi di laboratorio . Chimicamente, è un polifenolo e un benzochinone .
Occorrenze
Atromentin è stato trovato in colture di Clitocybe subilludens e in estratti di Hydnellum peckii . I primi enzimi nella sua biosintesi sono stati caratterizzati in Tapinella panuoides . Uno di questi è chiamato atromentin sintetasi .
Attività biologiche
Un certo numero di in vitro attivi biologici di atromentin sono stati studiati. Atromentin possiede attività antibatterica in vitro , inibendo l'enzima enoil-acil vettore proteina reduttasi (essenziale per la biosintesi degli acidi grassi ) nel batterio Streptococcus pneumoniae . Atromentin ha dimostrato di essere uno stimolante della muscolatura liscia . Inoltre induce l' apoptosi nelle cellule U937 isolate di leucemia umana . È anche un anticoagulante .
Basi genetiche ed enzimatiche dell'atromentina
Atromentin è catalizzato da due unità di acido 4-idrossifenilpiruvico (4-HPP) tramite un enzima peptide sintetasi nonribosomiale (atromentin sintetasi), contenente l'architettura del dominio adenilazione-tiolazione-tioesterasi (AT-TE). 4-HPP è prodotto da una deaminazione tramite un'aminotransferasi. La base genetica di questi due geni è raggruppata (cioè adiacente l'una all'altra). Questi enzimi sono stati caratterizzati per la prima volta in Tapinella panuoides sovraesprimendo i rispettivi geni (AtrA e AtrD) in E. coli e incubando l'oloenzima con 4-HPP per osservare la formazione di atromentina. Questa è stata seguita dalla caratterizzazione dell'enzima GreA in Suillus grevillei , sei (InvA1-6, di cui InvA1, 2 e 5 erano funzionali) in Paxillus involutus e NPS3 da Serpula lacrymans . Inoltre, esiste un altro gene adiacente e conservato che codifica per una alcol deidrogenasi / ossidoreduttasi la cui funzione non è chiara. Nella maggior parte dei casi i geni biosintetici raggruppati si trovano ortologhi nei basidiomiceti. Un motivo promotore comune è stato trovato condiviso tra l'atromentina sintetasi e l'aminotransferasi di 23 diversi basidiomiceti produttori di atromentina che era in quasi tutti i casi assente dall'alcol deidrogenasi, indicando la co-regolazione dei due geni essenziali che assicurano la produzione di atromentina da un fattore di trascrizione comune . Ulteriori motivi promotori sono stati identificati prima dei geni atromentin per ectomycorrihzae che erano assenti dai marciumi bruni, indicando una regolazione genetica dissimile dell'atromentino. I geni per l'atromentin sintetasi e l'aminotransferasi di S. lacrymans sono stati sovraregolati durante la co-incubazione con i batteri.
Codice amminoacidico nonribosomiale per la biosintesi
L'enzima peptide sintetasi nonribosomiale (atromentin sintetasi) che condensa simmetricamente due monomeri di 4-HPP ha un dominio di adenilazione che accetta i substrati prima della catalisi. Il dominio accettore contiene un codice di 10 aminoacidi noto come codice nonribosomiale (codice NRPS). Qui viene utilizzato l'esempio dell'atromentin sintetasi di Suills grevillei , GreA. Il codice si trova nelle posizioni degli amminoacidi 235 (V), 236 (A), 239 (E), 278 (F), 299 (S), 301 (G), 322 (G), 320 (A), 331 ( C), 517 (K). Il codice si allinea con atromentin sintetasi da S. lacrymans (NPS3), Tapinella panuoides (AtrA) e Paxillus involutus (InvAs). Allo stesso modo, il codice NRPS per la produzione di atromentin supporta il codice universale per altri composti aromatici derivati da alfa-chetoacido, come quelli da L -fenilalanina come ralfuranone B tramite acido fenilpiruvico e da L- triptofano come didemetil asterricinone D tramite indolo-3- acido piruvico (si noti che l'atromentino deriva dall'aromatico alfa-cheto acido L-tirosina tramite l'acido 4-idrossifenilpiruvico).
Per InvAs da Paxillus involutus , è stato trovato anche un motivo amminoacidico comune nel dominio della tioesterasi (ultimo dominio) che supportava i dati biochimici dell'enzima che era funzionale o meno per completare la formazione di atromentina.
Biosintesi dell'atromentina
L'amminoacido aromatico L- tirosina è il precursore dell'acido 4-idrossifenilpiruvico e 2 unità di 4-HPP vengono condensate per formare atromentina. La fase iniziale è la deaminazione tramite un'aminotransferasi. La seconda fase è catalizzata da un enzima simile alla peptide sintetasi nonribosomiale (simile a NRPS, perché non ha un dominio di condensazione canonico, chiamato atromentina / chinone sintetasi). Il dominio di adenilazione di questo enzima simile a NRPS accetta 4-HPP come determinato dal test di scambio ATP-PPi. L'enzima, quando prodotto in E. coli , deve essere innescato nella sua forma olo tramite una fosfopanteteinil transferasi (Ppant), sebbene l'E. Coli possa innescare in vivo l'apoenzima (ad esempio tramite EntD). Le mutande sono state utilizzate con successo da cDNA derivato da A. nidulans (es. NpgA), Streptomyces verticillus (Svp) e Paxillus involutus (PptA). Alcuni studi, in particolare sul batterio Burkholderia thailandensis di Biggins et al., Hanno dimostrato che il gene dell'aminotransferasi può essere assente e questa attività può essere fornita tramite il suo metabolismo primario.
Pigmenti Congener
L'atromentin è il precursore di vari altri pigmenti. Pulvinic acido tipo famiglia includono acido variegatic , acido xerocomic , acido homoxerocomic , acido isoxerocomic , acido atromentic , variegatorubin , xerocomorubin , e altri derivati modificati. I principali pigmenti di tipo acido pulvinico sono stati trovati secreti durante la co-incubazione con batteri o l'introduzione a un contenuto di azoto organico elevato (rispetto alla crescita su un mezzo non induttore contenente azoto inorganico). I diarilciclopentenoni includono involutina, involuton, girocicanina, giroporina (variante ossidata della girocianina), anidroinvolutina e camonixina. Sebbene strutturalmente simili, le grevilline (AD) derivano dalla 4-HPP, il precursore dell'atromentino. Le grevilline sono un marker chemiotassonomico per il genere Suillus. Le modifiche di atromentin includono leucoatromentin, leucomentin-3, leucomentin-4 e cylcoleucomelone. Inoltre, l'acido eleporico è un derivato che proviene dal clade thelephoroid. I vari enzimi coinvolti nella formazione di questi pigmenti a parte la base genetica ed enzimatica per la produzione del suo precursore atromentino sono sconosciuti.
Biosintesi ridondante
In Paxillus involutus , sei enzimi peptidici sintetasi nonribosomiali sono stati identificati nel genoma annotato disponibile tramite il portale JGI MycoCosm. Questi geni, denominati InvA1,2,3,4,5 e 6, erano sovraespressi in E. coli e i geni sono stati caratterizzati co-incubando l'apoenzima con 4-HPP per determinare la formazione di atromentina come notato dalla sua caratteristica Spettro UV-Vis e massa monoisotopica. Tre dei sei enzimi sono risultati funzionali. Ciò ha mostrato una ridondanza senza precedenti per la produzione di atromentin in un basidiomicete.
