Virus dell'epatite B -Hepatitis B virus
| Virus dell'epatite B | |
|---|---|

|
|
| Microscopia elettronica a trasmissione microscopio che mostra virus dell'epatite B virioni | |
|
Classificazione dei virus |
|
| (non classificato): | Virus |
| regno : | riboviria |
| Regno: | Pararnavira |
| Filo: | Artverviricota |
| Classe: | Revtraviriceti |
| Ordine: | Blubervirales |
| Famiglia: | Hepadnaviridae |
| Genere: | ortoepadnavirus |
| Specie: |
Virus dell'epatite B
|
Il virus dell'epatite B ( HBV ), è un virus a DNA parzialmente a doppio filamento, una specie del genere Orthohepadnavirus e un membro dellafamiglia di virus Hepadnaviridae . Questo virus causa la malattia epatite B .
Malattia
Nonostante esista un vaccino per prevenire l'epatite B, l'HBV rimane un problema di salute globale. L'epatite B può essere acuta e successivamente diventare cronica, portando ad altre malattie e condizioni di salute. Oltre a causare epatite, l'infezione da HBV può portare a cirrosi e carcinoma epatocellulare .
È stato anche suggerito che potrebbe aumentare il rischio di cancro al pancreas .
Ruoli nella malattia
L'infezione virale da virus dell'epatite B (HBV) provoca molti cambiamenti negli epatociti dovuti all'azione diretta di una proteina codificata dal virus, HBx , e cambiamenti indiretti dovuti a un grande aumento delle specie reattive dell'ossigeno intracellulare (ROS) dopo l'infezione. L'HBx sembra disregolare un certo numero di vie cellulari. L'HBx provoca in parte la disregolazione legandosi al DNA genomico , modificando i modelli di espressione dei miRNA, influenzando le metiltransferasi dell'istone, legandosi alla proteina SIRT1 per attivare la trascrizione e cooperando con le metilasi e le demetilasi dell'istone per modificare i modelli di espressione cellulare. L'HBx è in parte responsabile dell'aumento di circa 10.000 volte dei ROS intracellulari in caso di infezione cronica da HBV. L'aumento dei ROS può essere causato, in parte, dalla localizzazione di HBx nei mitocondri dove HBx diminuisce il potenziale di membrana mitocondriale. Inoltre, un'altra proteina dell'HBV , l' HBsAg , aumenta anche i ROS attraverso le interazioni con il reticolo endoplasmatico .
L'aumento delle specie reattive dell'ossigeno (ROS) dopo l'infezione da HBV provoca infiammazione, che porta ad un ulteriore aumento dei ROS. I ROS causano più di 20 tipi di danni al DNA. Il danno ossidativo al DNA è mutageno. Inoltre, la riparazione del danno al DNA può causare alterazioni epigenetiche nel sito del danno durante la riparazione del DNA. Alterazioni e mutazioni epigenetiche possono causare difetti nel macchinario cellulare che poi contribuiscono alla malattia epatica . Quando l'accumulo di cambiamenti epigenetici e mutazionali alla fine causa la progressione verso il cancro , le alterazioni epigenetiche sembrano avere un ruolo maggiore in questa carcinogenesi rispetto alle mutazioni. Solo uno o due geni, TP53 e forse ARID1A , sono mutati in più del 20% dei tumori epatici mentre 41 geni hanno promotori ipermetilati (che reprimono l'espressione genica ) in più del 20% dei tumori epatici, con sette di questi geni ipermetilati in oltre il 75% dei tumori del fegato. Oltre alle alterazioni nei siti di riparazione del DNA , le alterazioni epigenetiche sono anche causate dall'HBx che recluta gli enzimi del DNA metiltransferasi , DNMT1 e/o DNMT3A , a specifici loci genici per alterarne i livelli di metilazione e l'espressione genica. HBx altera anche l'acetilazione dell'istone che può influenzare l'espressione genica.
Diverse migliaia di geni codificanti proteine sembrano avere siti di legame per HBx. Oltre ai geni codificanti proteine, anche circa 15 microRNA e 16 RNA lunghi non codificanti sono influenzati dal legame di HBx ai loro promotori. Ogni microRNA alterato può influenzare l'espressione di diverse centinaia di RNA messaggeri (vedi microRNA ).
Classificazione
Il virus dell'epatite B è classificato nel genere Orthohepadnavirus , che contiene altre 11 specie. Il genere è classificato come parte della famiglia Hepadnaviridae , che contiene altri quattro generi, Avihepadnavirus , Herpetohepadnavirus , Metahepadnavirus e Parahepadnavirus . Questa famiglia di virus è l'unico membro dell'ordine virale Blubervirales . Virus simili all'epatite B sono stati trovati in tutte le scimmie ( oranghi , gibboni , gorilla e scimpanzé ), nelle scimmie del Vecchio Mondo e nelle scimmie lanose del Nuovo Mondo (il virus dell'epatite B delle scimmie lanose ), suggerendo un'origine antica per questo virus nei primati .
Il virus è suddiviso in quattro principali sierotipi (adr, adw, ayr, ayw) basati su epitopi antigenici presenti sulle sue proteine dell'involucro . Questi sierotipi si basano su un determinante comune (a) e due coppie di determinanti che si escludono a vicenda (d/y e w/r). I ceppi virali sono stati inoltre suddivisi in dieci genotipi (A–J) e quaranta sottogenotipi in base alla variazione complessiva della sequenza nucleotidica del genoma. I genotipi hanno una distribuzione geografica distinta e vengono utilizzati per tracciare l'evoluzione e la trasmissione del virus. Le differenze tra i genotipi influenzano la gravità della malattia, il decorso e la probabilità di complicanze e la risposta al trattamento ed eventualmente alla vaccinazione. I sierotipi e i genotipi non corrispondono necessariamente.
Il genotipo D ha 10 sottogenotipi.
Specie non classificate
Un certo numero di specie simili all'epatite B non ancora classificate sono state isolate dai pipistrelli.
Morfologia
Struttura
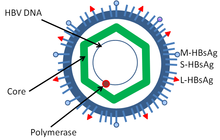
Il virus dell'epatite B è un membro della famiglia degli Hepadnavirus . La particella virale, chiamata particella Dane ( virion ), è costituita da un involucro lipidico esterno e da un nucleo nucleocapside icosaedrico composto da proteine . Il nucleocapside racchiude il DNA virale e una DNA polimerasi che ha un'attività di trascrittasi inversa simile ai retrovirus. L'involucro esterno contiene proteine incorporate che sono coinvolte nel legame virale e nell'ingresso nelle cellule sensibili. Il virus è uno dei più piccoli virus animali con involucro con un diametro del virione di 42 nm, ma esistono forme pleomorfe , compresi corpi filamentosi e sferici privi di nucleo. Queste particelle non sono infettive e sono composte dal lipide e dalla proteina che fa parte della superficie del virione, chiamato antigene di superficie ( HBsAg ), e viene prodotto in eccesso durante il ciclo di vita del virus.
Componenti
Esso consiste in:
- HBsAg ( antigene di superficie dell'epatite B ) è stata la prima proteina del virus dell'epatite B ad essere scoperta. Consiste di proteine piccole (S), medie (M) e grandi (L).
- HBcAg (antigene core dell'epatite B) è la principale proteina strutturale del nucleocapside icosaedrico dell'HBV e ha funzione di replicazione del virus . La formazione del capside è il principale fattore di infezione della cellula. L'HBcAg contribuisce alla clearance dell'HBV in vivo , ma non è noto se l'HBcAg debba essere nella forma del capside per contribuire alla clearance virale.
- La DNA polimerasi del virus dell'epatite B è incorporata nel nucleocapside insieme all'RNA pregenomico (pgRNA). All'interno del capside, il pgRNA subisce la trascrizione inversa, formando il filamento di DNA (-). Allo stesso tempo, la maggior parte dello stampo di RNA viene degradata dall'attività RNasi della polimerasi. Questo è seguito dalla sintesi del filamento di DNA (+) e la polimerasi finisce per legarsi covalentemente al filamento di DNA (-). La polimerasi viene scartata dopo che il virione ha infettato una nuova cellula.
- HBeAg (antigene dell'involucro dell'epatite B) può essere trovato tra il nucleo del nucleocapside icosaedrico e l'involucro lipidico, ma è considerato "non particolato" ed è secreto e si accumula nel siero. HBeAg e HBcAg sono realizzati dallo stesso telaio di lettura .
- L'HBx è piccolo, lungo 154 aminoacidi , non strutturale e ha un ruolo importante nella malattia epatica associata all'HBV e nella replicazione dell'HBV nelle cellule HepG2 . Molte attività sono state collegate all'espressione di HBx. Tuttavia, i meccanismi molecolari di molte di queste attività sono sconosciuti. Questa proteina è multifunzionale e attiva le vie di segnalazione cellulare ed è essenziale per l' infezione virale .
Il virus dell'epatite D richiede che le particelle dell'involucro dell'HBV diventino virulente.
Evoluzione
L'evoluzione precoce dell'epatite B, come quella di tutti i virus, è difficile da stabilire.
La divergenza di orthohepadnavirus e avihepadnavirus si è verificata circa 125.000 anni fa (intervallo del 95% 78.297-313.500). Sia l'Avihepadnavirus che l'Orthohepadna hanno iniziato a diversificarsi circa 25.000 anni fa. La ramificazione in questo momento porta all'emergere dei genotipi Orthohepadna A–H. I ceppi umani hanno un antenato comune più recente che risale a 7.000 (intervallo 95%: 5.287–9.270) a 10.000 (intervallo 95%: 6.305–16.681) anni fa.
L'Avihepadnavirus manca di una proteina X, ma nel genoma dell'hepadnavirus dell'anatra è presente un frame di lettura dell'X vestigiale. La proteina X potrebbe essersi evoluta da una DNA glicosilasi .
Il tasso di mutazioni non sinonime in questo virus è stato stimato in circa 2 × 10 -5 amminoacidi per sito all'anno. Il numero medio di sostituzioni nucleotidiche/sito/anno è ~7,9 × 10 -5 .
Una seconda stima dell'origine di questo virus suggerisce che un antenato comune più recente dei ceppi umani si sia evoluto circa 1500 anni fa. Il più recente antenato comune dei ceppi aviari è stato collocato a 6000 anni fa. Il tasso di mutazione è stato stimato in ~ 10-6 sostituzioni/sito/anno.
Un'altra analisi con un set di dati più ampio suggerisce che l'epatite B ha infettato gli esseri umani 33.600 anni fa (densità posteriore superiore del 95% 22.000-47.100 anni fa. Il tasso di sostituzione stimato era di 2,2 × 10 -6 sostituzioni/sito/anno. Un aumento significativo della popolazione è stata notata negli ultimi 5.000 anni L'infezione incrociata di specie tra oranghi e gibboni si è verificata negli ultimi 6.100 anni.
L'esame delle sequenze nel diamante mandarino ha riportato l'origine di questo genere almeno a 40 milioni di anni fa e forse a 80 milioni di anni fa . Le specie di scimpanzé , gorilla , oranghi e gibboni si raggruppano con isolati umani. Le specie non primate includevano il virus dell'epatite marmotta, il virus dell'epatite dello scoiattolo terrestre e il virus dell'epatite dello scoiattolo artico. Sono state descritte anche numerose specie che infettano i pipistrelli. È stato proposto che una specie di pipistrello del Nuovo Mondo possa essere l'origine della specie di primati.
Uno studio sugli isolati dalla popolazione umana dell'Artico circumpolare ha proposto che l'antenato del sottogenotipo B5 (il tipo endemico trovato in questa popolazione) che il virus ancestrale abbia avuto origine in Asia circa 2000 anni fa (95% HPD 900 aC - 830 dC). La coalescenza avvenne intorno al 1000 d.C. Questo sottogenotipo si è diffuso dall'Asia inizialmente alla Groenlandia e poi si è diffuso verso ovest negli ultimi 400 anni.
La più antica evidenza di infezione da epatite B risale all'età del bronzo . Le prove sono state ottenute da resti umani di 4.500 anni. Secondo lo studio del 2018, i genomi virali ottenuti dal sequenziamento shotgun sono diventati i più antichi mai recuperati da campioni di vertebrati. È stato anche scoperto che alcuni antichi ceppi virali dell'epatite infettano ancora gli esseri umani, mentre altri si sono estinti. Ciò ha smentito la credenza che l'epatite B abbia avuto origine nel Nuovo Mondo e si sia diffusa in Europa intorno al XVI secolo.
genoma
Taglia
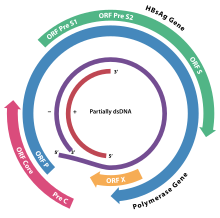
Il genoma dell'HBV è costituito da DNA circolare , ma è insolito perché il DNA non è completamente a doppia elica. Un'estremità del filamento a tutta lunghezza è collegata alla DNA polimerasi virale . Il genoma è lungo 3020-3320 nucleotidi (per il filamento a lunghezza intera) e lungo 1700-2800 nucleotidi (per il filamento corto).
Codifica
Il filamento di senso negativo (non codificante) è complementare all'mRNA virale. Il DNA virale si trova nel nucleo subito dopo l'infezione della cellula. Il DNA parzialmente a doppio filamento viene reso completamente a doppio filamento mediante il completamento del filamento di senso (+) da parte delle DNA polimerasi cellulari (la DNA polimerasi virale viene utilizzata per uno stadio successivo) e la rimozione della proteina della polimerasi virale (P) dal (- ) filamento senso e una breve sequenza di RNA dal filamento senso (+). Le basi non codificanti vengono rimosse dalle estremità del filamento di senso (-) e le estremità vengono riunite.
I geni virali sono trascritti dalla RNA polimerasi II cellulare nel nucleo cellulare da un modello di DNA circolare covalentemente chiuso (cccDNA). Nel genoma dell'HBV sono stati identificati due enhancer denominati enhancer I (EnhI) e enhancer II (EnhII). Entrambi i potenziatori mostrano una maggiore attività nelle cellule di origine epatica e insieme guidano e regolano l'espressione delle trascrizioni virali complete. Ci sono quattro geni conosciuti codificati dal genoma chiamati C, P, S e X. La proteina core è codificata dal gene C (HBcAg), e il suo codone di inizio è preceduto da un codone di inizio AUG in-frame a monte da cui il viene prodotta la proteina pre-core. L'HBeAg è prodotto dall'elaborazione proteolitica della proteina pre-core. La DNA polimerasi è codificata dal gene P. Il gene S è il gene che codifica per l'antigene di superficie (HBsAg). Il gene HBsAg è un lungo frame di lettura aperto ma contiene tre codoni di "inizio" (ATG) in frame che dividono il gene in tre sezioni, pre-S1, pre-S2 e S. A causa dei codoni di inizio multipli, polipeptidi di tre vengono prodotte diverse dimensioni chiamate grandi, medie e piccole (pre-S1 + pre-S2 + S, pre-S2 + S o S). La funzione della proteina codificata dal gene X non è completamente compresa, ma alcune prove suggeriscono che potrebbe funzionare come transattivatore trascrizionale.
Diversi elementi di RNA non codificanti sono stati identificati nel genoma dell'HBV. Questi includono: HBV PREalpha , HBV PREbeta e HBV RNA segnale epsilon di incapsidazione .
genotipi
I genotipi differiscono di almeno l'8% della sequenza e hanno distribuzioni geografiche distinte e questo è stato associato alla storia antropologica. All'interno dei genotipi sono stati descritti sottotipi: questi differiscono del 4-8% del genoma.
Ci sono otto genotipi conosciuti etichettati da A a H.
È stato descritto un possibile nuovo genotipo "I", ma l'accettazione di questa notazione non è universale.
Da allora sono stati riconosciuti altri due genotipi. L'elenco corrente (2014) ora va da A a J. Sono riconosciuti anche diversi sottotipi.
Ci sono almeno 24 sottotipi.
Diversi genotipi possono rispondere al trattamento in modi diversi.
- Genotipi individuali
Il tipo F che diverge dagli altri genomi del 14% è il tipo più divergente conosciuto. Il tipo A è prevalente in Europa , Africa e sud-est asiatico , comprese le Filippine . I tipi B e C sono predominanti in Asia ; il tipo D è comune nell'area mediterranea, in Medio Oriente e in India ; il tipo E è localizzato nell'Africa subsahariana; il tipo F (o H) è limitato all'America centrale e meridionale . Il tipo G è stato trovato in Francia e Germania . I genotipi A, D e F sono predominanti in Brasile e tutti i genotipi si verificano negli Stati Uniti con frequenze dipendenti dall'etnia.
I ceppi E e F sembrano aver avuto origine rispettivamente nelle popolazioni aborigene dell'Africa e del Nuovo Mondo.
Il tipo A ha due sottotipi: Aa (A1) in Africa/Asia e Filippine e Ae (A2) in Europa/Stati Uniti.
Il tipo B ha due distribuzioni geografiche distinte: Bj/B1 ('j'—Giappone) e Ba/B2 ('a'—Asia). Il tipo Ba è stato ulteriormente suddiviso in quattro cladi (B2-B4).
Il tipo C ha due sottotipi geograficamente: Cs (C1) nel sud-est asiatico e Ce (C2) nell'Asia orientale. I sottotipi C sono stati suddivisi in cinque cladi (C1-C5). Un sesto clade (C6) è stato descritto nelle Filippine ma solo in un isolato fino ad oggi. Il tipo C1 è associato a Vietnam , Myanmar e Thailandia ; tipo C2 con Giappone , Corea e Cina ; tipo C3 con Nuova Caledonia e Polinesia ; C4 con l' Australia ; e C5 con le Filippine . Un ulteriore sottotipo è stato descritto in Papua , Indonesia .
Il tipo D è stato suddiviso in 7 sottotipi (D1-D7).
Il tipo F è stato suddiviso in 4 sottotipi (F1-F4). La F1 è stata ulteriormente suddivisa in 1a e 1b. In Venezuela i sottotipi F1, F2 e F3 si trovano negli amerindi orientali e occidentali. Tra i sudamericani è stata trovata solo la F3. I sottotipi Ia, III e IV presentano una distribuzione geografica ristretta (rispettivamente America Centrale, Nord e Sud del Sud America) mentre i cladi Ib e II si trovano in tutte le Americhe eccetto rispettivamente nel Nord America del Sud e del Nord America.
Ciclo vitale
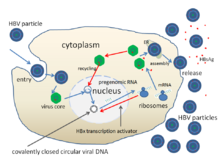
Il ciclo di vita del virus dell'epatite B è complesso. L'epatite B è uno dei pochi virus non retrovirali conosciuti che utilizzano la trascrizione inversa come parte del suo processo di replicazione.
- allegato
- Il virus ottiene l'ingresso nella cellula legandosi ai recettori sulla superficie della cellula ed entrandovi per endocitosi mediata dalla clatrina o dalla caveolina-1 . L'HBV si lega inizialmente al proteoglicano dell'eparina solfato . Il segmento pre-S1 della proteina HBV L si lega quindi strettamente al polipeptide cotrasportatore del sodio taurocolato (NTCP) del recettore della superficie cellulare , codificato dal gene SLC10A1 . NTCP si trova principalmente nella membrana sinusoidale delle cellule epatiche . La presenza di NTCP nelle cellule epatiche è correlata alla specificità tissutale dell'infezione da HBV.
- Penetrazione
- Dopo l'endocitosi, la membrana del virus si fonde con la membrana della cellula ospite, rilasciando il nucleocapside nel citoplasma.
- non rivestimento
- Poiché il virus si moltiplica tramite l'RNA prodotto da un enzima ospite, il DNA genomico virale deve essere trasferito al nucleo cellulare. Si pensa che il capside venga trasportato sui microtubuli al poro nucleare . Le proteine del nucleo si dissociano dal DNA virale parzialmente a doppio filamento, che viene quindi reso completamente a doppio filamento (dalle DNA polimerasi dell'ospite) e trasformato in DNA circolare covalentemente chiuso ( cccDNA ) che funge da modello per la trascrizione di quattro mRNA virali .
- replica
- L'mRNA più grande, (che è più lungo del genoma virale), viene utilizzato per creare le nuove copie del genoma e per produrre la proteina core del capside e la DNA-polimerasi RNA-dipendente virale .
- Assemblea
- Queste quattro trascrizioni virali subiscono un'ulteriore elaborazione e vanno a formare virioni di progenie che vengono rilasciati dalla cellula o restituiti al nucleo e riciclati per produrre ancora più copie.
- Pubblicazione
- Il lungo mRNA viene quindi trasportato di nuovo nel citoplasma dove la proteina virione P sintetizza il DNA tramite la sua attività di trascrittasi inversa.
Geni transattivati
HBV ha la capacità di transattivare FAM46A .
Guarda anche
- Vaccino contro l'epatite B
- Analoghi nucleosidici
- Oncovirus (virus del cancro)
Riferimenti