miR-155 - miR-155
| pre-mir-155 | |
|---|---|
 struttura secondaria pre-mir-155 e conservazione della sequenza.ard
| |
| Identificatori | |
| Simbolo | miR-155 |
| Rfam | RF00731 |
| famiglia miRBase | MIPF0000157 |
| Altri dati | |
| tipo di RNA | microRNA |
| Dominio/i | Eucariota ; |
| Strutture PDB | PDBe |
| MIR155 | |||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Identificatori | |||||||||||||||||||||||||
| Alias | MIR155 , MIRN155, miRNA155, mir-155, miR-155, microRNA 155 | ||||||||||||||||||||||||
| ID esterni | OMIM : 609337 GeneCard : MIR155 | ||||||||||||||||||||||||
| |||||||||||||||||||||||||
| Ortologhi | |||||||||||||||||||||||||
| Specie | Umano | Topo | |||||||||||||||||||||||
| Entrez |
|
||||||||||||||||||||||||
| Ensembl |
|
||||||||||||||||||||||||
| UniProt |
|
||||||||||||||||||||||||
| RefSeq (mRNA) |
|
|
|||||||||||||||||||||||
| RefSeq (proteine) |
|
|
|||||||||||||||||||||||
| Posizione (UCSC) | Chr 21: 25.57 – 25.57 Mb | n / A | |||||||||||||||||||||||
| Ricerca PubMed | n / A | ||||||||||||||||||||||||
| Wikidata | |||||||||||||||||||||||||
| |||||||||||||||||||||||||
MiR-155 è un microRNA che nell'uomo è codificata dal MIR155 ospite gene o MIR155HG . MiR-155 svolge un ruolo in vari processi fisiologici e patologici . Il controllo molecolare esogeno in vivo dell'espressione di miR-155 può inibire la crescita maligna , le infezioni virali e migliorare la progressione delle malattie cardiovascolari .
Scoperta
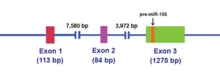
Il MIR155HG è stato inizialmente identificato come un gene che è stato attivato trascrizionalmente dall'inserimento del promotore in un comune sito di integrazione retrovirale nei linfomi a cellule B ed era precedentemente chiamato BIC (cluster di integrazione delle cellule B). Il MIR155HG viene trascritto dalla RNA polimerasi II e il risultante RNA di ~ 1.500 nucleotidi viene incapsulato e poliadenilato. Il miR-155 a singolo filamento di 23 nucleotidi, che è ospitato nell'esone 3, viene successivamente elaborato dalla molecola di RNA genitore.
biogenesi
La trascrizione dell'RNA MIR155HG non contiene un lungo frame di lettura aperto (ORF), tuttavia include un anello di stelo accoppiato in modo imperfetto che viene conservato tra le specie. Questo RNA non codificante ( ncRNA ) è ora definito come miRNA primario (pri-miRNA). Una volta miR-155 pri-miRNA viene trascritto, questa trascrizione viene scisso dal nucleare complesso microprocessore , i cui componenti principali sono il tipo RNasi III endonucleasi Drosha e 8 DiGeorge regione critica ( DGCR8 proteine), per produrre un nucleotide 65 stem- miRNA precursore del ciclo (pre-mir-155) (vedi Figura 2).
Dopo l'esportazione dal nucleo da parte dell'exportin-5, le molecole pre-mir-155 vengono scisse vicino all'ansa terminale da Dicer con conseguente duplex di RNA di ~ 22 nucleotidi. Dopo la scissione di Dicer, una proteina Argonaute (Ago) si lega ai brevi duplex di RNA, formando il nucleo di un complesso multi-subunità chiamato complesso di silenziamento indotto dall'RNA ( RISC ). In modo simile ai duplex di siRNA , uno dei due filamenti, il "miRNA passeggero" (miR-155*), viene rilasciato e degradato mentre l'altro filamento, denominato "filo guida" o "miRNA maturo" (miR-155 ), viene mantenuto all'interno del RISC.
Dati recenti suggeriscono che entrambi i bracci della forcina pre-miRNA possono dare origine a miRNA maturi. A causa del numero crescente di esempi in cui due miRNA maturi funzionali vengono elaborati da bracci opposti dello stesso pre-miRNA, i prodotti pre-mir-155 sono ora indicati con il suffisso -5p (dal braccio 5') (ad es. miR-155 -5p) e -3p (dal braccio 3′) (es. miR-155-3p) dopo il loro nome (vedi Figura 3).
Una volta che miR-155-5p/-3p è assemblato nel RISC, queste molecole riconoscono successivamente il loro RNA messaggero bersaglio ( mRNA ) mediante interazioni di accoppiamento di basi tra i nucleotidi 2 e 8 di miR-155-5p/-3p (la regione seme) e nucleotidi complementari prevalentemente nella regione 3'-non tradotta ( 3'-UTR ) degli mRNA (vedi Figura 4 e 5 sotto). Infine, con il miR-155-5p/-3p che agisce come un adattatore per il RISC, gli mRNA legati al complesso sono soggetti a repressione traduzionale (cioè inibizione dell'inizio della traduzione ) e/o degradazione dopo deadenilazione.
Conservazione evolutiva
Le prime analisi filogenetiche hanno dimostrato che la sequenza di pre-mir-155 e miR-155-5p era conservata tra uomo, topo e pollo. Recenti dati di sequenziamento annotati hanno scoperto che 22 diversi organismi tra cui mammiferi, anfibi, uccelli, rettili, ascidie e lamprede di mare, esprimono un miR-155-5p conservato. [1] Attualmente sono disponibili molti meno dati di sequenza relativi al miR-155-3p, quindi non è chiaro quanto sia conservato questo miRNA tra le specie. [2]
Distribuzione dei tessuti
L' analisi Northern blot ha rilevato che il pri-miRNA di miR-155 era abbondantemente espresso nella milza e nel timo umani e rilevabile nel fegato, nei polmoni e nei reni. Successivamente, esperimenti di reazione a catena della polimerasi ( PCR ) hanno dimostrato che miR-155-5p era rilevabile in tutti i tessuti umani studiati. L'analisi della sequenza di librerie di cloni di piccoli RNA confrontando l'espressione di miRNA con tutti gli altri sistemi di organi esaminati ha stabilito che miR-155-5p era uno dei cinque miRNA (cioè miR-142, miR-144, miR-150, miR-155 e miR-223 ) che è specifica per le cellule ematopoietiche comprese cellule B , cellule T , monociti e granulociti . Insieme, questi risultati suggeriscono che miR-155-5p è espresso in numerosi tessuti e tipi di cellule e, pertanto, può svolgere un ruolo critico in un'ampia varietà di processi biologici, inclusa l' emopoiesi.
Sebbene pochissimi studi abbiano studiato i livelli di espressione di miR-155-3p, Landgraf et al. stabilito che i livelli di espressione di questo miRNA erano molto bassi nelle cellule ematopoietiche. Inoltre, le analisi PCR hanno scoperto che mentre miR-155-3p era rilevabile in un certo numero di tessuti umani, i livelli di espressione di questo miRNA erano 20-200 volte inferiori rispetto ai livelli di miR-155-5p. Anche se la funzione di miR-155-3p è stata ampiamente ignorata, diversi studi ora suggeriscono che, in alcuni casi (astrociti e cellule dendritiche plasmacitoidi), sia miR-155-5p che -3p possono essere maturati funzionalmente da pre-mi- 155.
obiettivi
L'analisi bioinformatica utilizzando TargetScan 6.2 (data di rilascio giugno 2012) [3] ha rivelato che esistono almeno 4.174 bersagli putativi di mRNA miR-155-5p umani, con un totale di 918 siti conservati (cioè tra topo e uomo) e 4.249 siti scarsamente conservati ( cioè solo umano). Sebbene l'algoritmo TargetScan 6.2 non possa essere utilizzato per determinare i bersagli putativi miR-155-3p, si potrebbe ipotizzare che questo miRNA possa anche potenzialmente regolare l'espressione di migliaia di bersagli mRNA.
Recentemente è stato assemblato un elenco completo di bersagli di miR-155-5p/mRNA che sono stati autenticati sperimentalmente sia dalla dimostrazione della regolazione della trascrizione endogena da parte di miR-155-5p sia dalla convalida della sequenza di semi di miR-155-5p attraverso un test reporter. Questo elenco includeva 140 geni e includeva proteine regolatrici per mielopoiesi e leucemogenesi (es. SHIP-1 , AICDA , ETS1 , JARID2 , SPI1 , ecc.), infiammazione (es. BACH1 , FADD , IKBKE , INPP5D , MYD88 , RIPK1 , SPI1 , SOCS . ecc.) e noti oncosoppressori (es. CEBPβ , IL17RB , PCCD4 , TCF12 , ZNF652 , ecc.). Il sito di legame convalidato miR-155-5p ospitato nell'mRNA SPI1 e il sito di legame convalidato miR-155-3p ospitato nell'mRNA IRAK3 sono mostrati rispettivamente nelle Figure 4 e 5.

Ruoli fisiologici
Ematopoiesi
L'emopoiesi è definita come la formazione e lo sviluppo di cellule del sangue, tutte derivate da cellule staminali ematopoietiche progenitrici (HSPC). Gli HSPC sono cellule primitive capaci di autorinnovarsi e inizialmente differenziarsi in cellule progenitrici mieloidi comuni (CMP) o cellule progenitrici linfoidi comuni (CLP). I CMP rappresentano la popolazione cellulare che è diventata la linea mieloide ed è il punto in cui inizia la mielopoiesi . Durante la mielopoiesi ha luogo un'ulteriore differenziazione cellulare che comprende la trombopoiesi , l' eritropoiesi , la granulopoiesi e la monocitopoiesi . CLP successivamente differenziarsi in cellule B e cellule T in un processo designato linfopoiesi . Dato che miR-155-5p è espresso nelle cellule ematopoietiche è stato ipotizzato che questo miRNA svolga un ruolo critico in questi processi di differenziazione cellulare. A sostegno di questa premessa, è stato scoperto che miR-155-5p è espresso in HSPC umani CD34(+) ed è stato ipotizzato che questo miRNA possa trattenere queste cellule in una fase precoce di progenitori staminali, inibendo la loro differenziazione in una forma più matura. cellula (cioè megacariocitica/eritroide/granulocitica/monocitica/linfoide B/linfoide T). Questa ipotesi è stata confermata quando gli HSPC trasdotti pre-mir-155 hanno generato 5 volte meno colonie mieloidi e 3 volte meno eritroidi. Inoltre, Hu et al. hanno dimostrato che la proteina homeobox, HOXA9 , regolava l' espressione di MIR155HG nelle cellule mieloidi e che questo miRNA svolgeva un ruolo funzionale nell'emopoiesi. Questi ricercatori hanno scoperto che l'espressione forzata di miR-155-5p nelle cellule del midollo osseo ha portato a una diminuzione del 50% circa di SPI1 (cioè PU.1), un fattore di trascrizione e un regolatore della mielopoiesi e un bersaglio convalidato di questo miRNA. È stato inoltre stabilito che la differenziazione in vitro delle cellule progenitrici eritroidi umane purificate ha determinato una progressiva diminuzione dell'espressione di miR-155-5p nei globuli rossi maturi. Inoltre, i topi carenti di pre-mir-155 hanno mostrato evidenti difetti nello sviluppo dei linfociti e nella generazione di risposte delle cellule B e T in vivo . Infine, è stato stabilito che lo sviluppo delle cellule T regolatorie ( Treg ) richiedeva miR-155-5p e questo miRNA ha dimostrato di svolgere un ruolo nell'omeostasi delle Treg e nella sopravvivenza globale prendendo di mira direttamente SOCS1 , un regolatore negativo per la segnalazione di IL-2 . Presi insieme, questi risultati suggeriscono fortemente che miR-155-5p è una molecola essenziale nel controllo di diversi aspetti dell'emopoiesi, tra cui mielopoiesi, eritropoiesi e linfopoiesi.
Sistema immune
Il sistema immunitario innato costituisce la prima linea di difesa contro gli agenti patogeni invasori ed è considerato il principale iniziatore delle risposte infiammatorie . La sua componente cellulare coinvolge principalmente monociti / macrofagi , granulociti e cellule dendritiche (DC), che vengono attivati al rilevamento di strutture patogene conservate ( PAMP ) da recettori di riconoscimento di pattern come i recettori Toll-like ((TLR)). L'espressione di MIR155HG (cioè miR-155-5p) è notevolmente migliorata dalla stimolazione dei macrofagi e delle cellule dendritiche con agonisti dei TLR. Poiché il lipopolisaccaride microbico (un agonista di TLR4) attiva una catena di eventi che portano alla stimolazione dei fattori di trascrizione NF-κB e AP-1 , è stato ipotizzato che l'attivazione dell'endotossina di MIR155HG possa essere mediata da tali fattori di trascrizione. Infatti, è stato scoperto che l'espressione di MIR155HG è attivata in cellule di macrofagi murini trattate con LPS (cioè Raw264.7) da un meccanismo mediato da NF-κB. Inoltre, l' infezione da H. pylori di macrofagi primari derivati dal midollo osseo murino ha provocato una up-regolazione dipendente da NF-κB di MIR155HG . Nel contesto dell'infezione virale da virus della stomatite vescicolare (VSV) è stato segnalato che la stimolazione dei macrofagi peritoneali murini provoca una sovraespressione di miR-155-5p attraverso una via dipendente dal gene I/JNK/NF-κB inducibile dall'acido retinoico. Il supporto per un ruolo di AP-1 nell'attivazione di MIR155HG proviene da studi che utilizzano stimoli rilevanti per l'infezione virale come il ligando TLR3 poly(I:C) o l' interferone beta (IFN-β). A valle di quegli stimoli AP-1 sembra giocare un ruolo importante nell'attivazione di MIR155HG .
Al suo inizio tramite l'attivazione, ad esempio, di TLR da parte di stimoli patogeni, miR-155-5p funziona come regolatore post-trascrizionale delle vie di segnalazione immunitaria innate. È importante sottolineare che miR-155-5p mostra una reattività simile agli stimoli dei patogeni (ad es. LPS agonista TLR4) come i principali mRNA marcatori pro-infiammatori. Una volta attivato, miR-155-5p sopprime i regolatori negativi dell'infiammazione. Questi includono inositolo polifosfato-5-fosfatasi (INPP5D denotato anche SHIP1) e soppressore della segnalazione di citochine 1 (SOCS1), la cui soppressione promuove la sopravvivenza cellulare, la crescita, la migrazione e le risposte anti-patogeno. Oltre a supportare l'attivazione delle vie di difesa, il miR-155-5p può anche limitare la forza della risposta infiammatoria dipendente da NF-kB risultante, suggerendo diverse funzioni di miR-155 nelle diverse fasi dell'infiammazione.
Prese insieme, queste osservazioni implicano che l'attivazione del MIR155HG può essere dipendente dal contesto dato che entrambi i meccanismi mediati da AP-1 e NF-κB regolano l'espressione di questo gene. Questi studi suggeriscono anche che un'ampia gamma di mediatori dell'infiammazione virale e batterica può stimolare l'espressione di miR-155-5p e indicare che esiste un'intima relazione tra infiammazione, immunità innata ed espressione di MIR155HG .
Attività e fenotipi
Ci sono prove che il miR-155 partecipa a cascate associate a malattie cardiovascolari e ipertensione, ed è stato anche scoperto che è implicato nell'immunità, instabilità genomica , differenziazione cellulare , infiammazione, infezioni associate a virus e cancro.
Ruoli protettivi di miR-155 possono sorgere in risposta alla sua azione sul silenziamento dei geni regolando così il loro tempo di espressione, le mutazioni nel sito bersaglio di miR-155 gli negano l'accesso ottimale necessario per provocare il silenziamento genico, portando a un'eccessiva abbondanza di attività delinquenti che possono go maligno , ad esempio, il ruolo di miR-155 come agente protettivo contro la predisposizione a tumori maligni associati alle cellule B è enfatizzato dal mantenimento dell'equilibrio dell'enzima citidina deaminasi ( AID ) indotto dall'attivazione . MiR-155 media la regolazione dell'abbondanza di AID e del tempo di espressione su segnali immunologici, tuttavia, le mutazioni nel bersaglio sull'mRNA dell'AID determinano la sua mancata risposta al silenziamento del miR-155 e portano all'espressione sfrenata della sua proteina causando picchi selvaggi di linfociti B immaturi e AID- traslocazioni cromosomiche mediate .
Significato clinico
Cardiovascolare
La trasfezione di miR-155 nei fibroblasti polmonari primari umani riduce l'espressione endogena della proteina AT1R del recettore dell'angiotensina II . Inoltre, AT1R media l'aumento della pressione sanguigna correlato all'angiotensina II e contribuisce alla patogenesi dell'insufficienza cardiaca. La funzione difettosa di miR-155 potrebbe essere implicata nell'ipertensione e nelle malattie cardiovascolari se il sito cis-regolatorio su 3` UTR di AT1R (sito bersaglio miR-155) fosse interessato a causa di un polimorfismo SNP nell'AT1R stesso. Questa mutazione è distruttiva del targeting di miR-155 e quindi preventiva della down-regulation dell'espressione di AT1R. Nella bassa pressione sanguigna la sovraespressione di miR-155 è correlata alla compromissione dell'attività di AT1R.
Immunità
miR-155 è coinvolto nell'immunità svolgendo ruoli chiave nella modulazione delle risposte immunitarie mediate da cellule umorali e innate, ad esempio, nei topi carenti di miR-155, la memoria immunologica è compromessa; facendolo cadere preda di attacchi ripetitivi di invasioni da parte dello stesso patogeno (Rodriguez et al. 2007), la maturazione e la specificità dei linfociti B carenti di miR-155 sono compromesse poiché il processo si basa sull'enzima AID che ha un bersaglio miR-155 in la sua estremità 3′ UTR. Le conseguenze fenotipiche che comportano la carenza di miR-155 nei topi si manifestano più avanti nella vita quando gli animali sviluppano lesioni polmonari e intestinali .
Le cellule B e T attivate mostrano un aumento dell'espressione di miR-155, lo stesso vale per i macrofagi e le cellule dendritiche del sistema immunitario . MiR-155 è fondamentale per il corretto sviluppo e maturazione dei linfociti. I dettagli delle varie manifestazioni dei livelli di miR-155 e il coinvolgimento in attività che accertano risposte immunitarie ottimali sono stati oggetto di molte ricerche:
Riduzione di IgG1
Cellule T e B difettose e risposte IgG1 marcatamente diminuite sono state osservate in topi con deficit di miR-155, IgG1 è ridotto mentre l'espressione dell'immunoglobulina IgM rimane normale in questi topi. Il cambiamento nei livelli di IgG1 può essere spiegato dal fatto che è un bersaglio per miR-155 nelle cellule B, l'mRNA che codifica per la proteina per la proteina Pu.1 del regolatore trascrizionale , l'elevazione della proteina Pu.1 predispone alla produzione difettosa di IgG1. Oltre a Pu.1, ci sono quasi 60 altri geni differenzialmente elevati nelle cellule B carenti di miR-155, un'ulteriore ispezione ha rivelato possibili siti bersaglio di miR-155 nelle regioni 3' UTR in questi geni.
Neoplasie linfocitarie
L' affinità e la specificità dei recettori maturi dei linfociti verso gli agenti patogeni sono alla base di risposte immunitarie adeguate, è necessaria una coordinazione ottimale del miR-155 per la produzione di linfociti B normali, la produzione di anticorpi ad alta affinità e il bilanciamento della segnalazione BCR. È stato dimostrato che il miR-155 può essere trasferito attraverso giunzioni gap da cellule leucemiche a cellule B sane e promuovere la loro trasformazione in cellule simili a tumori.
La selezione delle cellule B competenti avviene nel centro germinativo dove sono addestrate a differenziare le cellule del corpo rispetto agli antigeni estranei, competono per il riconoscimento dell'antigene e per l'aiuto delle cellule T, in questo modo di pressione selettiva quelle cellule B che hanno dimostrato recettori ad alta affinità e la cooperazione con le cellule T ( maturazione dell'affinità ) vengono reclutate e dispiegate nel midollo osseo o diventano cellule B di memoria, la terminazione apoptotica avviene per quelle cellule B che falliscono la competizione. Le cellule B immature che sono carenti di miR-155 eludono l'apoptosi a causa di elevati livelli di proteina Bcl-2 ; una proteina che è risultata coinvolta nei tumori maligni delle cellule B e controllata dal miR-155.
Infiammazione
Le risposte infiammatorie a fattori scatenanti come il TNF-α coinvolgono macrofagi con componenti che includono miR-155. miR-155 è sovraespresso nella dermatite atopica e contribuisce all'infiammazione cronica della pelle aumentando la risposta proliferativa delle cellule T(H) attraverso la downregulation di CTLA-4. Nelle malattie autoimmuni come l'artrite reumatoide, il miR-155 ha mostrato una maggiore espressione nei tessuti dei pazienti e nei fibroblasti sinoviali. Nella sclerosi multipla, è stata misurata anche una maggiore espressione di mir-155 nelle cellule mieloidi periferiche e residenti nel SNC, compresi i monociti del sangue circolante e la microglia attivata. È stato anche scoperto che il mir-155 è implicato nell'infiammazione. La sovraespressione di mir-155 porterà a uno stato infiammatorio cronico nell'uomo.
virus a DNA
Nei virus a DNA , i miRNA sono stati verificati sperimentalmente, i miRNA nei virus sono codificati da dsDNA, esempi di tali virus includono herpesvirus come Humans-Epstein-Barr Virus ( EBV ) e adenovirus , un altro virus che esprime miRNA simile a miR-155 nei polli è il oncogenico MDV-1 il cui parente non oncogeno MDV-2 non lo fa, questo suggerisce l'implicazione di miR-155 nella linfomagenesi. I virus possono sfruttare i miRNA dell'ospite nella misura in cui usano i miRNA dell'ospite per codificare per i cloni virali, ad esempio: miR-K12-11 nell'Herpesvirus associato al sarcoma di Kaposi ha una regione di specificità del bersaglio ortologa a quella dei miR-155; imitando l'azione di miR-155 e, condividendo bersagli con esso, quindi si può pensare di sopprimere l'accessibilità di miR-155 ai suoi bersagli per competizione e questo in effetti sottoregola l'espressione dei geni che giocano ruoli nella crescita cellulare e nell'apoptosi in un modo che sfida regolamenti da miR-155. EBV modula l'espressione del miR-155 dell'ospite, che è essenziale per la crescita delle cellule B infettate da EBV. Le cellule infettate da EBV hanno aumentato l'espressione di miR-155 disturbando così l'equilibrio di espressione per i geni che regolano la trascrizione in quelle cellule.
Cancro
Il silenziamento eccessivo di miR-155 può provocare l'attivazione di cascate oncogene che iniziano con la resistenza apoptotica, la proteina nucleare pro-apoptotica-53-indotta-proteina1 ( TP53INP1 ) è silenziata da miR-155, sovraespressione di miR-155 porta a livelli ridotti di TP53INP1 negli adenocarcinomi duttali pancreatici e possibilmente in altri tumori epiteliali in cui l'attività di TP53INP1 viene persa con conseguente evasione dell'apoptosi e attacchi di crescita incontrollati.
L'inattivazione del DNA Mismatch Repair ( MMR ) come identificato dall'aumento dei tassi di mutazione è la causa della sindrome di Lynch (LS), nota anche come cancro colorettale ereditario non poliposico (HNPCC), la down-regulation della proteina che controlla l'MMR viene effettuata dalla sovraespressione di miR-155, l'MMR è controllato da un gruppo di proteine conservate, la ridotta attività di queste proteine si traduce in elevati livelli di mutazioni nel fenotipo innescando una marcia verso lo sviluppo di questo tipo di cancro.
Altri tipi di tumori in cui è stata segnalata la sovraespressione di miR-155 includono: carcinoma della tiroide, cancro al seno, cancro del colon, cancro della cervice uterina e cancro del polmone, dove la quantificazione dei profili di espressione di miR-155 distinti può potenzialmente servire come segnali per il rilevamento e la valutazione del tumore dell'esito della prognosi. È dimostrato in un'analisi che l'espressione di miR-155 è associata alla sopravvivenza nel carcinoma mammario triplo negativo.
Appunti
Guarda anche
Riferimenti
Ulteriori letture
- Bentwich I, Avniel A, Karov Y, Aharonov R, Gilad S, Barad O, Barzilai A, Einat P, Einav U, Meiri E, Sharon E, Spector Y, Bentwich Z (luglio 2005). "Identificazione di centinaia di microRNA umani conservati e non conservati". Genetica della natura . 37 (7): 766-770. doi : 10.1038/ng1590 . PMID 15965474 . S2CID 7746954 .
- Huang RS, Hu GQ, Lin B, Lin ZY, Sun CC (dicembre 2010). "Il silenziamento del microRNA-155 migliora la risposta infiammatoria e l'assorbimento dei lipidi nei macrofagi THP-1 umani stimolati da lipoproteine a bassa densità ossidate". Giornale di medicina investigativa . 58 (8): 961–967. doi : 10.2310/jim.0b013e3181ff46d7 . PMID 21030878 . S2CID 83123221 .
- Ceolotto G, Papparella I, Bortoluzzi A, Strapazzon G, Ragazzo F, Bratti P, Fabricio AS, Squarcina E, Gion M, Palatini P, Semplicini A (feb 2011). "Interazione tra miR-155, polimorfismo AT1R A1166C ed espressione di AT1R in giovani ipertesi non trattati" . Giornale americano di ipertensione . 24 (2): 241–246. doi : 10.1038/ajh.2010.211 . PMID 20966899 .
- Wang G, Tam LS, Li EK, Kwan BC, Chow KM, Luk CC, Li PK, Szeto CC (dicembre 2010). "MiR-146a e MiR-155 privi di cellule sieriche e urinarie in pazienti con lupus eritematoso sistemico". Il giornale di reumatologia . 37 (12): 2516-2522. doi : 10.3899/jrheum.100308 . PMID 20952466 . S2CID 23208771 .
- Kutty RK, Nagineni CN, Samuel W, Vijayasarathy C, Hooks JJ, Redmond TM (novembre 2010). "Le citochine infiammatorie regolano l'espressione del microRNA-155 nelle cellule epiteliali del pigmento retinico umano attivando la via JAK/STAT" . Comunicazioni di ricerca biochimica e biofisica . 402 (2): 390-395. doi : 10.1016/j.bbrc.200.10.042 . PMC 2.992.362 . PMID 20950585 .
- Imaizumi T, Tanaka H, Tajima A, Yokono Y, Matsumiya T, Yoshida H, Tsuruga K, Aizawa-Yashiro T, Hayakari R, Inoue I, Ito E, Satoh K (2010). "IFN-γ e TNF-α inducono sinergicamente microRNA-155 che regola l'espressione di TAB2/IP-10 nelle cellule mesangiali umane". Giornale americano di nefrologia . 32 (5): 462–468. doi : 10.1159/000321365 . PMID 20948191 . S2CID 12859943 .
- Thompson RC, Herscovitch M, Zhao I, Ford TJ, Gilmore TD (gennaio 2011). "NF-kappaB down-regola l'espressione del marcatore del linfoma B CD10 attraverso una via miR-155/PU.1" . Il giornale di chimica biologica . 286 (3): 1675–1682. doi : 10.1074/jbc.M110.177063 . PMC 3023462 . PMID 20947507 .
- Wang P, Hou J, Lin L, Wang C, Liu X, Li D, Ma F, Wang Z, Cao X (novembre 2010). "Il feedback inducibile del microRNA-155 promuove la segnalazione dell'IFN di tipo I nell'immunità innata antivirale mirando al soppressore della segnalazione delle citochine 1" . Giornale di immunologia . 185 (10): 6226-6233. doi : 10.4049/jimmunol.1000491 . PMID 20937844 .
- O'Connell RM, Kahn D, Gibson WS, Round JL, Scholz RL, Chaudhuri AA, Kahn ME, Rao DS, Baltimora D (ottobre 2010). "MicroRNA-155 promuove l'infiammazione autoimmune migliorando lo sviluppo delle cellule T infiammatorie" . Immunità . 33 (4): 607–619. doi : 10.1016/j.immuni.2010.09.09 . PMC 2966521 . PMID 20888269 .
- Xia QS, Ishigaki Y, Zhao X, Shimasaki T, Nakajima H, Nakagawa H, Takegami T, Chen ZH, Motoo Y (gennaio 2011). "L'SMG-1 umano è coinvolto nella up-regulation primaria del microRNA-155/BIC indotta dalla gemcitabina nelle cellule PANC-1 del cancro del pancreas umano". Pancreas . 40 (1): 55-60. doi : 10.1097/MPA.0b013e3181e89f74 . PMID 20871480 . S2CID 39789659 .
- Zhou H, Huang X, Cui H, Luo X, Tang Y, Chen S, Wu L, Shen N (dicembre 2010). "miR-155 e il suo partner in forma stellare miR-155* regolano in modo cooperativo la produzione di interferone di tipo I da parte delle cellule dendritiche plasmacitoidi umane" . Sangue . 116 (26): 5885-5894. doi : 10.1182/sangue-2010-04-280156 . PMID 20852130 .
- Linnstaedt SD, Gottwein E, Skalsky RL, Luftig MA, Cullen BR (novembre 2010). "Il microRNA cellulare indotto viralmente miR-155 svolge un ruolo chiave nell'immortalità delle cellule B da parte del virus Epstein-Barr" . Giornale di virologia . 84 (22): 11670–11678. doi : 10.1128/JVI.01248-10 . PMC 2977875 . PMID 20844043 .
- Zheng L, Xu CC, Chen WD, Shen WL, Ruan CC, Zhu LM, Zhu DL, Gao PJ (ottobre 2010). "MicroRNA-155 regola l'espressione del recettore di tipo 1 dell'angiotensina II e la differenziazione fenotipica nei fibroblasti avventiziali vascolari". Comunicazioni di ricerca biochimica e biofisica . 400 (4): 483-488. doi : 10.1016/j.bbrc.2010.08.067 . PMID 20735984 .
- Tili E, Michaille JJ, Adair B, Alder H, Limagne E, Taccioli C, Ferracin M, Delmas D, Latruffe N, Croce CM (settembre 2010). "Il resveratrolo riduce i livelli di miR-155 sovraregolando il miR-663, un microRNA che prende di mira JunB e JunD" . Cancerogenesi . 31 (9): 1561–1566. doi : 10.1093/carcin/bgq143 . PMC 4.647.642 . PMID 20622002 .
- Boesch-Saadatmandi C, Loboda A, Wagner AE, Stachurska A, Jozkowicz A, Dulak J, Döring F, Wolffram S, Rimbach G (marzo 2011). "Effetto della quercetina e dei suoi metaboliti isorhamnetina e quercetina-3-glucuronide sull'espressione genica infiammatoria: ruolo di miR-155". Il giornale di biochimica nutrizionale . 22 (3): 293-299. doi : 10.1016/j.jnutbio.2010.02.008 . PMID 20579867 .
- Liu J, van Mil A, Vrijsen K, Zhao J, Gao L, Metz CH, Goumans MJ, Doevendans PA, Sluijter JP (luglio 2011). "MicroRNA-155 previene la morte delle cellule necrotiche nelle cellule progenitrici dei cardiomiociti umani attraverso il targeting RIP1" . Giornale di medicina cellulare e molecolare . 15 (7): 1474–1482. doi : 10.1111/j.1582-4934.2010.01104.x . PMC 3823192 . PMID 20550618 .
- Pogribny IP, Starlard-Davenport A, Tryndyak VP, Han T, Ross SA, Rusyn I, Beland FA (ottobre 2010). "La differenza nell'espressione dei microRNA epatici miR-29c, miR-34a, miR-155 e miR-200b è associata alla suscettibilità specifica del ceppo alla steatoepatite non alcolica alimentare nei topi" . Indagine di laboratorio . 90 (10): 1437–1446. doi : 10.1038/labinvest.2010.113 . PMC 4281935 . PMID 20548288 .
- Zhang Y, Diao Z, Su L, Sun H, Li R, Cui H, Hu Y (maggio 2010). "MicroRNA-155 contribuisce alla preeclampsia down-regolando CYR61". Giornale americano di ostetricia e ginecologia . 202 (5): 466.e1-7. doi : 10.1016/j.ajog.2010.01.057 . PMID 20452491 .
- Hu YL, Fong S, Largman C, Shen WF (settembre 2010). "HOXA9 regola il miR-155 nelle cellule ematopoietiche" . Ricerca sugli acidi nucleici . 38 (16): 5472-5478. doi : 10.1093/nar/gkq337 . PMC 2938212 . PMID 20444872 .
- McCoy CE, Sheedy FJ, Qualls JE, Doyle SL, Quinn SR, Murray PJ, O'Neill LA (luglio 2010). "IL-10 inibisce l'induzione di miR-155 da parte dei recettori toll-like" . Il giornale di chimica biologica . 285 (27): 20492-20498. doi : 10.1074/jbc.M110.102111 . PMC 2898307 . PMID 20435894 .
- Yin Q, Wang X, Fewell C, Cameron J, Zhu H, Baddoo M, Lin Z, Flemington EK (luglio 2010). "MicroRNA miR-155 inibisce la segnalazione della proteina morfogenetica ossea (BMP) e la riattivazione del virus Epstein-Barr mediata da BMP" . Giornale di virologia . 84 (13): 6318-6327. doi : 10.1128/JVI.00635-10 . PMC 2903268 . PMID 20427544 .
- Zhu J, Hu XQ, Guo GL, Zhang Y, Wang OC, You J, Huang QD, Zhang XH (febbraio 2010). "[Espressione e il suo significato clinico di miR-155 nel carcinoma mammario primario umano]". Zhonghua Wai Ke Za Zhi [Giornale cinese di chirurgia] . 48 (3): 205-208. PMID 20388420 .
- Kong W, He L, Coppola M, Guo J, Esposito NN, Coppola D, Cheng JQ (giugno 2010). "MicroRNA-155 regola la sopravvivenza cellulare, la crescita e la chemiosensibilità mirando a FOXO3a nel cancro al seno" . Il giornale di chimica biologica . 285 (23): 17869–17879. doi : 10.1074/jbc.M110.101055 . PMC 2878550 . PMID 20371610 .
- Xia QS, Ishigaki Y, Sun L, Chen R, Motoo Y (gennaio 2010). "[Effetto dei farmaci antitumorali sull'espressione di BIC/miR-155 nelle cellule PANC-1] del cancro del pancreas umano]". Zhonghua Yi Xue Za Zhi . 90 (2): 123-127. PMID 20356498 .
- Jiang S, Zhang HW, Lu MH, He XH, Li Y, Gu H, Liu MF, Wang ED (aprile 2010). "MicroRNA-155 funziona come un OncomiR nel cancro al seno prendendo di mira il soppressore del gene di segnalazione delle citochine 1" . Ricerca sul cancro . 70 (8): 3119-3127. doi : 10.1158/0008-5472.CAN-09-4250 . PMID 20354188 .
- Ryu JK, Hong SM, Karikari CA, Hruban RH, Goggins MG, Maitra A (2010). "L'espressione aberrante di MicroRNA-155 è un evento precoce nella progressione multistep dell'adenocarcinoma pancreatico" . Pancreatologia . 10 (1): 66-73. doi : 10.1159/000231984 . PMC 2865485 . PMID 20332664 .
- Fabani MM, Abreu-Goodger C, Williams D, Lyons PA, Torres AG, Smith KG, Enright AJ, Gait MJ, Vigorito E (luglio 2010). "Inibizione efficiente della funzione miR-155 in vivo da acidi nucleici peptidici" . Ricerca sugli acidi nucleici . 38 (13): 4466-4475. doi : 10.1093/nar/gkq160 . PMC 2910044 . PMID 20223773 .
- Tang B, Xiao B, Liu Z, Li N, Zhu ED, Li BS, Xie QH, Zhuang Y, Zou QM, Mao XH (aprile 2010). "Identificazione di MyD88 come nuovo bersaglio di miR-155, coinvolto nella regolazione negativa dell'infiammazione indotta da Helicobacter pylori". Lettere FEB . 584 (8): 1481–1486. doi : 10.1016/j.febslet.2010.02.063 . PMID 20219467 . S2CID 41984991 .
- Fassi Fehri L, Koch M, Belogolova E, Khalil H, Bolz C, Kalali B, Mollenkopf HJ, Beigier-Bompadre M, Karlas A, Schneider T, Churin Y, Gerhard M, Meyer TF (2010). "Helicobacter pylori induce miR-155 nelle cellule T in modo dipendente da cAMP-Foxp3" . PLOS UNO . 5 (3): e9500. Bibcode : 2010PLoSO...5.9500F . doi : 10.1371/journal.pone.0009500 . PMC 2830477 . PMID 20209161 .
- Sidorkiewicz M, Grek M, Jozwiak B, Majda-Stanislawska E, Piekarska A, Bartkowiak J (2010). "Espressione del precursore del microRNA-155 nelle cellule mononucleate del sangue periferico da pazienti con epatite C dopo trattamento antivirale". Acta Virologica . 54 (1): 75-78. doi : 10.4149/av_2010_01_75 . PMID 201617 .
- Gueta K, Molotski N, Gerchikov N, Mor E, Savion S, Fein A, Toder V, Shomron N, Torchinsky A (2010). "Alterazioni indotte da teratogeni nell'espressione di microRNA-34, microRNA-125b e microRNA-155: correlazione con genotipo p53 embrionale e fenotipo degli arti" . Biologia dello sviluppo BMC . 10 : 20. doi : 10.1186/1471-213X-10-20 . PMC 2841584 . PMID 20170545 .
- Rai D, Kim SW, McKeller MR, Dahia PL, Aguiar RC (febbraio 2010). "Il targeting di SMAD5 collega il microRNA-155 alla via del TGF-beta e alla linfomagenesi" . Atti della National Academy of Sciences degli Stati Uniti d'America . 107 (7): 3111-3116. Bibcode : 2010PNAS..107.3111R . doi : 10.1073/pnas.0910667107 . PMC 2.840.369 . PMID 20133617 .
- Cremer TJ, Ravneberg DH, Clay CD, Piper-Hunter MG, Marsh CB, Elton TS, Gunn JS, Amer A, Kanneganti TD, Schlesinger LS, Butchar JP, Tridandapani S (2009). "L'induzione di MiR-155 da parte di F. novicida, ma non del virulento F. tularensis, determina una down-regulation della SHIP e una maggiore risposta delle citochine pro-infiammatorie" . PLOS UNO . 4 (12): e8508. Bibcode : 2009PLoSO...4.8508C . doi : 10.1371/journal.pone.0008508 . PMC 2794384 . PMID 20041145 .
- Forrest AR, Kanamori-Katayama M, Tomaru Y, Lassmann T, Ninomiya N, Takahashi Y, de Hoon MJ, Kubosaki A, Kaiho A, Suzuki M, Yasuda J, Kawai J, Hayashizaki Y, Hume DA, Suzuki H (febbraio 2010 ). "L'induzione di microRNA, mir-155, mir-222, mir-424 e mir-503, promuove la differenziazione monocitica attraverso la regolazione combinatoria". Leucemia . 24 (2): 460-466. arXiv : 1007.2689 . doi : 10.1038/leu.2009.246 . PMID 19956200 . S2CID 2333658 .
- Pedersen IM, Otero D, Kao E, Miletic AV, Hother C, Ralfkiaer E, Rickert RC, Gronbaek K, David M (agosto 2009). "Onco-miR-155 mira a SHIP1 per promuovere la crescita TNFalfa-dipendente dei linfomi a cellule B" . EMBO Medicina Molecolare . 1 (5): 288-295. doi : 10.1002/emmm.200900028 . PMC 2771872 . PMID 19890474 .
- He M, Xu Z, Ding T, Kuang DM, Zheng L (ottobre 2009). "Il microRNA-155 regola la produzione di citochine infiammatorie nei macrofagi associati al tumore attraverso il targeting di C/EBPbeta" . Immunologia cellulare e molecolare . 6 (5): 343–352. doi : 10.1038/cmi.2009.45 . PMC 4.003.217 . PMID 19887047 .
- Tili E, Croce CM, Michaille JJ (2009). "miR-155: sulla diafonia tra infiammazione e cancro". Recensioni internazionali di immunologia . 28 (5): 264-284. doi : 10.1080/08830180903093796 . PMID 19811312 . S2CID 205589961 .
- Stahl HF, Fauti T, Ullrich N, Bopp T, Kubach J, Rust W, Labhart P, Alexiadis V, Becker C, Hafner M, Weith A, Lenter MC, Jonuleit H, Schmitt E, Mennerich D (2009). "L'inibizione del miR-155 sensibilizza le cellule CD4+ Th per la soppressione mediata da TREG" . PLOS UNO . 4 (9): e7158. Bibcode : 2009PLoSO...4.7158S . doi : 10.1371/journal.pone.0007158 . PMC 2743997 . PMID 19777054 .
- Bolisetty MT, Dy G, Tam W, Beemon KL (dicembre 2009). "Il ceppo T del virus della reticoloendoteliosi induce miR-155, che prende di mira JARID2 e promuove la sopravvivenza cellulare" . Giornale di virologia . 83 (23): 12009–12017. doi : 10.1128/JVI.01182-09 . PMC 2786729 . PMID 19759154 .
- Wang B, Majumder S, Nuovo G, Kutay H, Volinia S, Patel T, Schmittgen TD, Croce C, Ghoshal K, Jacob ST (ottobre 2009). "Ruolo del microRNA-155 nelle prime fasi dell'epatocarcinogenesi indotta da una dieta carente di colina e definita da aminoacidi in topi C57BL/6" . Epatologia . 50 (4): 1152-1161. doi : 10.1002/hep.23100 . PMC 2.757.532 . PMID 19711427 .
- Pottier N, Maurin T, Chevalier B, Puisségur MP, Lebrigand K, Robbe-Sermesant K, Bertero T, Lino Cardenas CL, Courcot E, Rios G, Fourre S, Lo-Guidice JM, Marcet B, Cardinaud B, Barbry P, Mari B (2009). "Identificazione del fattore di crescita dei cheratinociti come bersaglio del microRNA-155 nei fibroblasti polmonari: implicazione nelle interazioni epiteliale-mesenchimale" . PLOS UNO . 4 (8): e6718. Bibcode : 2009PLoSO...4.6718P . doi : 10.1371/journal.pone.0006718 . PMC 2726943 . PMID 19701459 .
- Xiao B, Liu Z, Li BS, Tang B, Li W, Guo G, Shi Y, Wang F, Wu Y, Tong WD, Guo H, Mao XH, Zou QM (settembre 2009). "Induzione di microRNA-155 durante l'infezione da Helicobacter pylori e il suo ruolo regolatorio negativo nella risposta infiammatoria" . Il giornale delle malattie infettive . 200 (6): 916–925. doi : 10.1086/605443 . PMID 19650740 .
- Worm J, Stenvang J, Petri A, Frederiksen KS, Obad S, Elmén J, Hedtjärn M, Straarup EM, Hansen JB, Kauppinen S (settembre 2009). "Il silenziamento del microRNA-155 nei topi durante la risposta infiammatoria acuta porta alla derepressione di c/ebp Beta e alla down-regulation del G-CSF" . Ricerca sugli acidi nucleici . 37 (17): 5784–5792. doi : 10.1093/nar/gkp577 . PMC 2761263 . PMID 19596814 .
- Costinean S, Sandhu SK, Pedersen IM, Tili E, Trotta R, Perrotti D, Ciarlariello D, Neviani P, Harb J, Kauffman LR, Shidham A, Croce CM (agosto 2009). "Src omologia 2 dominio contenente inositolo-5-fosfatasi e proteina beta legante il potenziatore CCAAT sono presi di mira da miR-155 nelle cellule B di topi transgenici Emicro-MiR-155" . Sangue . 114 (7): 1374–1382. doi : 10.1182/sangue-2009-05-220814 . PMC 2727407 . PMID 19520806 .
- Ruggiero T, Trabucchi M, De Santa F, Zupo S, Harfe BD, McManus MT, Rosenfeld MG, Briata P, Gherzi R (settembre 2009). "LPS induce l'elaborazione dipendente dalla proteina regolatoria di splicing di tipo KH dei precursori di microRNA-155 nei macrofagi". Giornale FASEB . 23 (9): 2898–2908. doi : 10.1096/fj.09-131342 . PMID 19423639 . S2CID 588328 .
- Martinez-Nunez RT, Louafi F, Friedmann PS, Sanchez-Elsner T (giugno 2009). "Il microRNA-155 modula la capacità di legame del patogeno delle cellule dendritiche (DC) mediante la down-regolazione della molecola di adesione intercellulare 3 specifica per le DC che afferra la non-integrina (DC-SIGN)" . Il giornale di chimica biologica . 284 (24): 16334–16342. doi : 10.1074/jbc.M109.011601 . PMC 2.713.543 . PMID 19386588 .
- O'Connell RM, Chaudhuri AA, Rao DS, Baltimora D (aprile 2009). "L'inositolo fosfatasi SHIP1 è un obiettivo primario di miR-155" . Atti della National Academy of Sciences degli Stati Uniti d'America . 106 (17): 7113-7118. Bibcode : 2009PNAS..106.7113O . doi : 10.1073/pnas.0902636106 . PMC 2678424 . PMID 19359473 .
- Kohlhaas S, Garden OA, Scudamore C, Turner M, Okkenhaug K, Vigorito E (marzo 2009). "Avanguardia: il target Foxp3 miR-155 contribuisce allo sviluppo delle cellule T regolatorie" . Giornale di immunologia . 182 (5): 2578-2582. doi : 10.4049/jimmunol.0803162 . PMID 19234151 .
- Ceppi M, Pereira PM, Dunand-Sauthier I, Barras E, Reith W, Santos MA, Pierre P (febbraio 2009). "MicroRNA-155 modula la via di segnalazione dell'interleuchina-1 in cellule dendritiche derivate da monociti umani attivati" . Atti della National Academy of Sciences degli Stati Uniti d'America . 106 (8): 2735–2740. Bibcode : 2009PNAS..106.2735C . doi : 10.1073/pnas.0811073106 . PMC 2650335 . PMID 19193853 .
- Habbe N, Koorstra JB, Mendell JT, Offerhaus GJ, Ryu JK, Feldmann G, Mullendore ME, Goggins MG, Hong SM, Maitra A (febbraio 2009). "MicroRNA miR-155 è un biomarcatore di neoplasia pancreatica precoce" . Biologia e terapia del cancro . 8 (4): 340-346. doi : 10.4161/cbt.8.4.7338 . PMC 2692997 . PMID 19106647 .
- Jung I, Aguiar RC (gennaio 2009). "Espressione e risultato di microRNA-155 nel linfoma diffuso a grandi cellule B" . Giornale britannico di ematologia . 144 (1): 138-140. doi : 10.1111/j.1365-2141.2008.07424.x . PMC 4654117 . PMID 19016736 .
- Romania P, Lulli V, Pelosi E, Biffoni M, Peschle C, Marziali G (nov 2008). "Il microRNA 155 modula la megacariopoiesi a livello di progenitori e precursori prendendo di mira i fattori di trascrizione Ets-1 e Meis1" . Giornale britannico di ematologia . 143 (4): 570-580. doi : 10.1111/j.1365-2141.2008.07382.x . PMID 18950466 . S2CID 9025761 .
- Zhao Y, Yao Y, Xu H, Lambeth L, Smith LP, Kgosana L, Wang X, Nair V (gennaio 2009). "Un funzionale MicroRNA-155 ortholog codificato dal virus della malattia di Marek oncogenico" . Giornale di virologia . 83 (1): 489-492. doi : 10.1128/JVI.01166-08 . PMC 2612317 . PMID 18945769 .
- Gatto G, Rossi A, Rossi D, Kroening S, Bonatti S, Mallardo M (Nov 2008). "La proteina 1 di membrana latente del virus di Epstein-Barr attiva la trascrizione di miR-155 attraverso la via NF-kappaB" . Ricerca sugli acidi nucleici . 36 (20): 6608-6619. doi : 10.1093/nar/gkn666 . PMC 2582607 . PMID 18940871 .
- Rahadiani N, Takakuwa T, Tresnasari K, Morii E, Aozasa K (dicembre 2008). "La proteina di membrana latente-1 del virus di Epstein-Barr induce l'espressione del cluster di integrazione delle cellule B, una forma precursore del microRNA-155, in linee cellulari di linfoma B". Comunicazioni di ricerca biochimica e biofisica . 377 (2): 579-583. doi : 10.1016/j.bbrc.2008.10.007 . PMID 18926796 .
- Kong W, Yang H, He L, Zhao JJ, Coppola D, Dalton WS, Cheng JQ (nov 2008). "Il microRNA-155 è regolato dalla via del fattore di crescita trasformante beta/Smad e contribuisce alla plasticità delle cellule epiteliali prendendo di mira RhoA" . Biologia molecolare e cellulare . 28 (22): 6773-6784. doi : 10.1128/MCB.00941-08 . PMC 2573297 . PMID 18794355 .
- Lu F, Weidmer A, Liu CG, Volinia S, Croce CM, Lieberman PM (nov 2008). "Il miR-155 indotto dal virus di Epstein-Barr attenua la segnalazione di NF-kappaB e stabilizza la persistenza del virus latente" . Giornale di virologia . 82 (21): 10436–10443. doi : 10.1128/JVI.00752-08 . PMC 2573162 . PMID 18753206 .
- Dorsett Y, McBride KM, Jankovic M, Gazumyan A, Thai TH, Robbiani DF, Di Virgilio M, Reina San-Martin B, Heidkamp G, Schwickert TA, Eisenreich T, Rajewsky K, Nussenzweig MC (maggio 2008). "MicroRNA-155 sopprime la traslocazione di Myc-Igh mediata dalla citidina deaminasi indotta dall'attivazione" . Immunità . 28 (5): 630-638. doi : 10.1016/j.immuni.2008.04.002 . PMC 2713656 . PMID 18455451 .
- Teng G, Hakimpour P, Landgraf P, Rice A, Tuschl T, Casellas R, Papavasiliou FN (maggio 2008). "MicroRNA-155 è un regolatore negativo della citidina deaminasi indotta dall'attivazione" . Immunità . 28 (5): 621–629. doi : 10.1016/j.immuni.2008.03.015 . PMC 2430982 . PMID 18450484 .
- Yin Q, McBride J, Fewell C, Lacey M, Wang X, Lin Z, Cameron J, Flemington EK (giugno 2008). "MicroRNA-155 è un gene indotto dal virus Epstein-Barr che modula i percorsi di espressione genica regolati dal virus Epstein-Barr" . Giornale di virologia . 82 (11): 5295-5306. doi : 10.1128/JVI.02380-07 . PMC 2395216 . PMID 18367535 .
- Zhang T, Nie K, Tam W (settembre 2008). "BIC viene elaborato in modo efficiente a microRNA-155 nelle cellule di linfoma di Burkitt" . Leucemia . 22 (9): 1795–1797. doi : 10.1038/leu.2008.62 . PMID 18354490 .
- Wang M, Tan LP, Dijkstra MK, van Lom K, Robertus JL, Harms G, Blokzijl T, Kooistra K, van T'veer MB, Rosati S, Visser L, Jongen-Lavrencic M, Kluin PM, van den Berg A ( maggio 2008). "Analisi dei miRNA nella leucemia linfatica cronica a cellule B: centri di proliferazione caratterizzati da bassa espressione di miR-150 e alta BIC/miR-155". Il Giornale di Patologia . 215 (1): 13-20. doi : 10.1002/percorso.2333 . PMID 18348159 . S2CID 29470560 .
- O'Connell RM, Rao DS, Chaudhuri AA, Boldin MP, Taganov KD, Nicoll J, Paquette RL, Baltimora D (marzo 2008). "L'espressione prolungata di microRNA-155 nelle cellule staminali ematopoietiche provoca un disturbo mieloproliferativo" . Il giornale di medicina sperimentale . 205 (3): 585-594. doi : 10.1084/jem.20072108 . PMC 2275382 . PMID 18299402 .
- Rai D, Karanti S, Jung I, Dahia PL, Aguiar RC (febbraio 2008). "Espressione coordinata di microRNA-155 e geni bersaglio previsti nel linfoma diffuso a grandi cellule B" . Genetica del cancro e citogenetica . 181 (1): 8-15. doi : 10.1016/j.cancergencyto.2007.10.008 . PMC 2276854 . PMID 18262046 .
- Gottwein E, Mukherjee N, Sachse C, Frenzel C, Majoros WH, Chi JT, Braich R, Manoharan M, Soutschek J, Ohler U, Cullen BR (dicembre 2007). "Un microRNA virale funziona come un ortologo del miR-155 cellulare" . Natura . 450 (7172): 1096-1099. Bibcode : 2007Natur.450.1096G . doi : 10.1038/nature05992 . PMC 2614920 . PMID 18075594 .
- Vigorito E, Perks KL, Abreu-Goodger C, Bunting S, Xiang Z, Kohlhaas S, Das PP, Miska EA, Rodriguez A, Bradley A, Smith KG, Rada C, Enright AJ, Toellner KM, Maclennan IC, Turner M ( dicembre 2007). "microRNA-155 regola la generazione di plasmacellule a commutazione di classe di immunoglobuline" . Immunità . 27 (6): 847-859. doi : 10.1016/j.immuni.2007.10.009 . PMC 4135426 . PMID 18055230 .
- Yin Q, Wang X, McBride J, Fewell C, Flemington E (febbraio 2008). "L'attivazione del recettore delle cellule B induce l'espressione di BIC/miR-155 attraverso un elemento AP-1 conservato" . Il giornale di chimica biologica . 283 (5): 2654-2662. doi : 10.1074/jbc.M708218200 . PMC 2810639 . PMID 18048365 .
- Tili E, Michaille JJ, Cimino A, Costinean S, Dumitru CD, Adair B, Fabbri M, Alder H, Liu CG, Calin GA, Croce CM (ottobre 2007). "Modulazione dei livelli di miR-155 e miR-125b a seguito di stimolazione lipopolisaccaride/TNF-alfa e loro possibili ruoli nella regolazione della risposta allo shock da endotossina" . Giornale di immunologia . 179 (8): 5082-5089. doi : 10.4049/jimmunol.179.8.5082 . PMID 17911593 .
- Gironella M, Seux M, Xie MJ, Cano C, Tomasini R, Gommeaux J, Garcia S, Nowak J, Yeung ML, Jeang KT, Chaix A, Fazli L, Motoo Y, Wang Q, Rocchi P, Russo A, Gleave M , Dagorn JC, Iovanna JL, Carrier A, Pébusque MJ, Dusetti NJ (ottobre 2007). "L'espressione della proteina nucleare 1 indotta dalla proteina tumorale 53 è repressa da miR-155 e il suo ripristino inibisce lo sviluppo del tumore pancreatico" . Atti della National Academy of Sciences degli Stati Uniti d'America . 104 (41): 16170–16175. Bibcode : 2007PNAS..10416170G . doi : 10.1073/pnas.0703942104 . PMC 2042180 . PMID 17911264 .
- Skalsky RL, Samols MA, Plaisance KB, Boss IW, Riva A, Lopez MC, Baker HV, Renne R (dicembre 2007). "Herpesvirus associato al sarcoma di Kaposi codifica un ortologo di miR-155" . Giornale di virologia . 81 (23): 12836–12845. doi : 10.1128/JVI.01804-07 . PMC 2169101 . PMID 17881434 .
- Sethupathy P, Borel C, Gagnebin M, Grant GR, Deutsch S, Elton TS, Hatzigeorgiou AG, Antonarakis SE (agosto 2007). "Il microRNA umano-155 sul cromosoma 21 interagisce in modo differenziale con il suo bersaglio polimorfico nella regione non tradotta AGTR1 3′: un meccanismo per polimorfismi funzionali a singolo nucleotide legati ai fenotipi" . Giornale americano di genetica umana . 81 (2): 405-413. doi : 10.1086/519979 . PMC 1950808 . PMID 17668390 .
- Martin MM, Buckenberger JA, Jiang J, Malana GE, Nuovo GJ, Chotani M, Feldman DS, Schmittgen TD, Elton TS (agosto 2007). "Il recettore di tipo 1 dell'angiotensina II umana +1166 A/C polimorfismo attenua il legame del microRNA-155" . Il giornale di chimica biologica . 282 (33): 24262-24269. doi : 10.1074/jbc.M701050200 . PMC 2413065 . PMID 17588946 .
- Rodriguez A, Vigorito E, Clare S, Warren MV, Couttet P, Soond DR, van Dongen S, Grocock RJ, Das PP, Miska EA, Vetrie D, Okkenhaug K, Enright AJ, Dougan G, Turner M, Bradley A (apr 2007). "Requisito di bic/microRNA-155 per la normale funzione immunitaria" . Scienza . 316 (5824): 608-611. Bibcode : 2007Sci...316..608R . doi : 10.1126/science.1139253 . PMC 2610435 . PMID 17463290 .
- Thai TH, Calado DP, Casola S, Ansel KM, Xiao C, Xue Y, Murphy A, Frendewey D, Valenzuela D, Kutok JL, Schmidt-Supprian M, Rajewsky N, Yancopoulos G, Rao A, Rajewsky K (aprile 2007) . "Regolazione della risposta del centro germinale da microRNA-155". Scienza . 316 (5824): 604-608. Bibcode : 2007Sci...316..604T . doi : 10.1126/science.1141229 . PMID 17463289 . S2CID 8174458 .
- O'Connell RM, Taganov KD, Boldin MP, Cheng G, Baltimora D (gennaio 2007). "MicroRNA-155 è indotto durante la risposta infiammatoria dei macrofagi" . Atti della National Academy of Sciences degli Stati Uniti d'America . 104 (5): 1604–1609. Bibcode : 2007PNAS..104.1604O . doi : 10.1073/pnas.0610731104 . PMC 1780072 . PMID 17242365 .
- Martin MM, Lee EJ, Buckenberger JA, Schmittgen TD, Elton TS (luglio 2006). "MicroRNA-155 regola l'espressione del recettore di tipo 1 dell'angiotensina II umana nei fibroblasti" . Il giornale di chimica biologica . 281 (27): 18277–18284. doi : 10.1074/jbc.M601496200 . PMID 16675453 .
- Chung KH, Hart CC, Al-Bassam S, Avery A, Taylor J, Patel PD, Vojtek AB, Turner DL (2006). "Vettori di espressione della RNA polimerasi II policistronica per l'interferenza dell'RNA basata su BIC/miR-155" . Ricerca sugli acidi nucleici . 34 (7): e53. doi : 10.1093/nar/gkl143 . PMC 1435982 . PMID 16614444 .
- Tam W, Dahlberg JE (febbraio 2006). "miR-155/BIC come microRNA oncogenico". Geni, cromosomi e cancro . 45 (2): 211-212. doi : 10.1002/gcc.20282 . PMID 16252262 . S2CID 8513721 .
- Kluiver J, Haralambieva E, de Jong D, Blokzijl T, Jacobs S, Kroesen BJ, Poppema S, van den Berg A (febbraio 2006). "Mancanza di espressione BIC e microRNA miR-155 nei casi primari di linfoma di Burkitt". Geni, cromosomi e cancro . 45 (2): 147-153. doi : 10.1002/gcc.20273 . PMID 16235244 . S2CID 16897980 .
- Jiang J, Lee EJ, Schmittgen TD (gennaio 2006). "Aumentata espressione di microRNA-155 nel virus di Epstein-Barr trasformato linee cellulari linfoblastoidi". Geni, cromosomi e cancro . 45 (1): 103-106. doi : 10.1002/gcc.20264 . PMID 16175574 . S2CID 19138209 .
- Kluiver J, Poppema S, de Jong D, Blokzijl T, Harms G, Jacobs S, Kroesen BJ, van den Berg A (ottobre 2005). "BIC e miR-155 sono altamente espressi in Hodgkin, linfomi primari mediastinici e diffusi a grandi cellule B". Il Giornale di Patologia . 207 (2): 243-249. doi : 10.1002/percorso.1825 . PMID 16041695 . S2CID 14329069 .
- Eis PS, Tam W, Sun L, Chadburn A, Li Z, Gomez MF, Lund E, Dahlberg JE (marzo 2005). "Accumulo di miR-155 e BIC RNA nei linfomi a cellule B umane" . Atti della National Academy of Sciences degli Stati Uniti d'America . 102 (10): 3627-3632. Bibcode : 2005PNAS..102.3627E . doi : 10.1073/pnas.050613102 . PMC 552785 . PMID 15738415 .
- Metzler M, Wilda M, Busch K, Viehmann S, Borkhardt A (febbraio 2004). "Alta espressione del precursore microRNA-155/BIC RNA nei bambini con linfoma di Burkitt". Geni, cromosomi e cancro . 39 (2): 167-169. doi : 10.1002/gcc.10316 . PMID 14695998 . S2CID 10009892 .
link esterno
- miRBase
- Voci miRBase per tutti i mir-155 conosciuti
- Pagina per la famiglia di precursori di microRNA mir-155 su Rfam